Provhantering, lysering och metoder för renframställning av nukleinsyra
Till innehållsförteckningen för Referensmetodik:Molekylärbiologisk diagnostik
Nukleinsyrapåvisning: Provhantering, lysering och metoder för renframställning av nukleinsyra[redigera]
Allmänt[redigera]
De molekylära amplifieringsmetodernas frammarsch har inneburit en revolution för mikrobiologisk diagnostik. Mycket kraft har lagts på att optimera metodernas känslighet, specificitet och snabbhet. Många av analysmetoderna kan detektera enskilda målsekvenser av eftersökta mikroorganismer inom 0,5 till 2,5 timmar. En förutsättning för en lyckad molekylärbiologisk analys är att hela analysprocessen fungerar. Det spelar ingen roll hur effektiv en amplifieringsmetod är om nukleinsyran i provmaterialet har degraderats. Detsamma gäller om man inte lyckas extrahera nukleinsyran i tillräcklig mängd eller renhet. En välfungerande preanalytisk process, som innefattar optimal provtagning, korrekt hantering av provet och effektiv nukleinsyraextraktion, är därför grundläggande för en känslig och specifik detektionsmetod.
Nukleinsyrastabilisering[redigera]
DNA är generellt sett en stabil molekyl medan RNA är mer instabil. Detta speglar de olika nukleinsyrornas funktioner i celler. DNA borgar för att den genetiska informationen bibehålls och förs vidare till nästa generation, medan RNA oftast är en kort budbärare av geninformation i översättningen från DNA till proteiner.
Även om DNA är stabilt så förstörs det i närvaro av deoxyribonukleaser (DNaser), som är DNA-nedbrytande nukleaser i bakterier och virus. För att DNA ska bibehålla sin stabilitet i ett kliniskt prov måste alltså DNaserna renas bort eller inaktiveras. De flesta DNaser är beroende av Mg2+-joner och inaktiveras av kelatorer som citrat eller EDTA. Fragmenterat DNA är känsligare för DNaser än intakt DNA, varför man generellt rekommenderar att renat fragmenterat DNA långtidsförvaras vid –20°C medan cirkulärt plasmid-DNA kan kylförvaras i Tris-EDTA buffert. Intorkat DNA är betydligt stabilare än DNA upplöst i vätska.
Enzymer som bryter ner RNA, ribonukleaser (RNaser), finns i princip överallt i omgivningen. RNaserna är betydligt svårare att inaktivera än DNaser. Sammantaget betyder detta att om man vill detektera RNA i ett prov så måste man stabilisera RNA och inaktivera RNaser. Om man inte har möjlighet att omedelbart extrahera RNA i provet bör det frysas vid –70°C eller stabiliseras genom tillsats av RNas-hämmande substanser. Provmaterial där man stabiliserat RNA kan förvaras i kyl (eller t o m i rumstemperatur) i väntan på extraktion. Viralt RNA skyddas i viss mån av viruskapsiden och ribosomalt RNA av sin sekundärstruktur, vilket gör att dessa RNA-former är mer stabila än mRNA och därmed tål en kortare tids kylförvaring innan extraktion. Även efter extraktion är RNA instabilt varför det bör långtidsförvaras vid –70°C eller kallare.
Nukleinsyraextraktion[redigera]
Allmänt[redigera]
Nukleinsyra förekommer i princip aldrig fritt i ett patientprov utan måste extraheras från celler eller viruspartiklar. Extraktionen bygger på två hörnstenar; lysering av celler eller viruspartiklar samt renframställning av nukleinsyran.Vid lyseringen av cellerna eller viruspartiklarna frigörs även nukleaser och andra cellkomponenter som bör elimineras eller inaktiveras för att inte störa den efterföljande metodiken för påvisning och analys. Vilken extraktionsmetod man väljer beror på en rad olika faktorer. Den ideala extraktionsmetoden ger ett bra utbyte av intakt nukleinsyra med hög renhet, från alla mikroorganismer och provtyper. Någon sådan metod finns inte i praktiken, utan extraktionsmetoden får anpassas utifrån de tekniska och analytiska krav man ställer på den givna analysmetoden.
Lyseringsmetoder[redigera]
Det finns många olika sätt att lysera celler eller viruspartiklar för att frigöra dess nukleinsyra (tabell 1). Ofta använder man kombinationer av lyseringsmetoder, men vid god tillgång på nukleinsyra och om analysmetoden är robust kan man nöja sig med att använda en fysikalisk lyseringmetod. För att undersöka om gramnegativa bakteriekolonier har en viss gen, räcker det oftast med upphettning i samband med PCR. Denna strategi fungerar dock inte för alla gramnegativa bakterier eftersom vissa har ett värmestabilt DNas som inte inaktiveras genom kokning. För grampositiva bakterier räcker oftast heller inte kokning som lyseringsmetod. I stället måste enzym tillsättas som bryter ned den kraftiga cellväggen eller ytproteiner för att bakterierna ska lysera. Filamentösa svampar är ännu mer rigida vilket innebär att deras cellväggar behöver spräckas på mekanisk väg genom homogenisering i närvaro av små glaskulor eller sonikering.
Man skall vara medveten om att nukleaser inte inaktiveras av lyseringsmetoderna som listas i tabell 1. Om inaktivering är önskvärd måste den fysikaliska lyseringsmetoden kombineras med en annan metod som inaktiverar nukleaser. Alternativt kan man använda en metod som snabbt eliminerar nukleaserna från den extraherade nukleinsyran.
Tabell 1. Exempel på metoder som används vid olika typer av cell-lysering

De kemiska lyseringsmetoderna kan användas ensamma eller i kombination med andra lyseringsmetoder. Detergenter används för att lösa upp cellmembran, hydrofoba proteiner och fetthaltigt provmaterial. Osmotiska metoder kan användas för att eliminera röda blodkroppar innan nukleinsyrainnehållande celler lyseras eller för att lysera bakterier vars cellvägg har gjorts instabil genom enzymatisk behandling.
Det klassiska sättet att på kemisk väg extrahera DNA är att använda en fenol/kloroformextraktion av provmaterial. För svåra provtyper som exempelvis biopsier startar man extraktionen genom lys med hjälp av detergenten SDS och proteinas K för att fortsätta med lys och denaturering genom tillsats av fenol/kloroform till extraktet. Med denna typ av lysering får man också en rening genom fas-separation som innebär att nukleinsyran och andra kolhydrater hamnar i vattenfasen. Lipider hamnar i den organiska fasen och denaturerade proteiner hamnar i interfasen mellan de båda.
Fenol/kloroformextraktion används sällan i dag eftersom de organiska lösningsmedlen är skadliga för både användarna och miljön. Man har numera gått över till att huvudsakligen använda kaotropa ämnen (denaturerande ämnen som guanidin-isotiocyanat eller guanidin-hydroklorid), för att lysera prov och extrahera DNA. Detta är ett mycket effektivt lyseringssätt som dessutom inaktiverar och denaturerar de flesta proteiner, inklusive nukleaser. För RNA preparation används ofta en kombination av guanidin-isotiocyanat och fenol/kloroformextraktion.
Metoder för renframställning av nukleinsyra[redigera]
När cellerna lyserats och nukleinsyra frigjorts, har samtidigt också ett antal oönskade komponenter frigjorts. Dessa bör helst elimineras innan extraktet används för analys. För att få maximal känslighet är det av värde att samtidigt koncentrera extraktet. Genom att precipitera (fälla) nukleinsyran med etanol eller isopropanol i närvaro av salter kan en hög koncentrationsgrad uppnås. Renhetsgraden vid alkoholfällning är dock inte tillräcklig så därför används alkoholprecipitation oftast som ett slutsteg efter exempelvis fenol/kloroformextraktion.
Det vanligaste sättet att rena och koncentrera nukleinsyra från cellextrakt är numera med kiseldioxidmatriser (silika-matriser) eller anjonbytare som båda med hög affinitet binder nukleinsyra. Genom att använda en buffert med en given jonstyrka och pH kan man selektivt binda nukleinsyra medan föroreningar i form av proteiner, polysackarider och lipider kan tvättas bort. Matriserna är bundna till någon form av kolonn, som förenklar tvättsteg och eluering av den rena nukleinsyran, eller till paramagnetiska kulor. Eftersom DNA och RNA skiljer sig åt kemiskt och fysikaliskt, kan man genom att variera pH och jonstyrka i tvätt- och elueringsbuffertar selektivt rena fram olika typer, eller längder, av nukleinsyra.
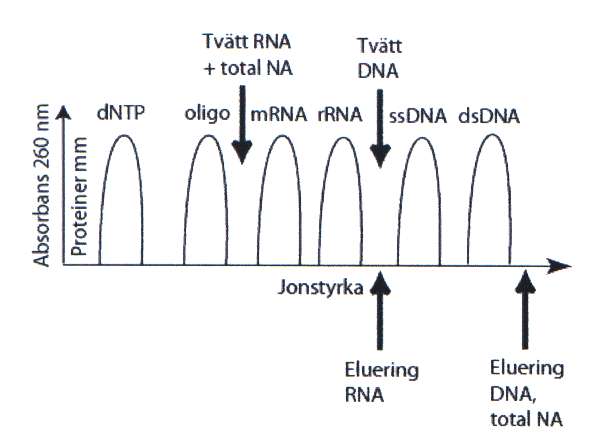
Dubbelsträngat DNA binder starkt till anjonbytarmatriser medan RNA binder svagare. Korta enkelsträngade DNA-oligonukleotider och nukleotider (dNTPs) binder ännu svagare vilket gör att de blir lätta att tvätta bort (figur 5).
Figur 5. Elueringsprofil från en anjonbytarmatris. Bild: Håkan Janson]]
För att eluera all nukleinsyra genomsköljs anjonbytaren direkt med en buffert med hög jonstyrka, medan man för att selektivt eluera RNA använder en buffert som har lägre jonstyrka än det som krävs för att eluera DNA, som då sitter kvar på matrisen. Vill man bara eluera DNA tvättar man matrisen med den jonstyrka som eluerar RNA, men inte DNA, för att sedan eluera med den högre jonstyrka som eluerar DNA. Eftersom eluatet från jonbytarmatriser har hög salthalt måste man precipitera nukleinsyran med alkohol och efter centrifugering suspendera pelleten i en lösning med lägre jonstyrka.
Kiseldioxidmatriser fungerar närmast omvänt jämfört med anjonbytare, eftersom nukleinsyra binder starkt till kiseldioxid vid hög jonstyrka och svagt vid låg jonstyrka. Här binder man alltså in och tvättar nukleinsyran till kiseldioxid med en buffert med hög jonstyrka. Man kan också tvätta med etanol eller aceton för att få bort lipider och torka ut matrisen innan nukleinsyran elueras med vatten eller med en buffert med låg jonstyrka. Detta ger en fördel eftersom eluatet kan användas direkt till analys utan att man behöver avsalta det.
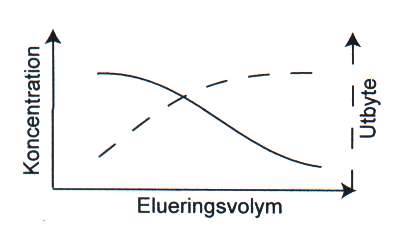
Genom att använda en elueringsvolym som är mindre än ursprungsvolymen på extraktet kan man koncentrera nukleinsyran vid elueringen. Utbytet är dock beroende av hur stor elueringsvolym man använder (figur 6). En stor elueringsvolym ger ett bra utbyte men med låg koncentration, medan en liten elueringsvolym ger ett koncentrerat eluat men med sämre utbyte. En nackdel med silika-matriser är att korta nukleinsyrafragment kan vara svåra att eluera oavsett vilken elueringsvolym man använder.
Figur 6. Koncentration och utbyte av nukleinsyra i förhållande till elueringsvolym. Bild: Håkan Janson
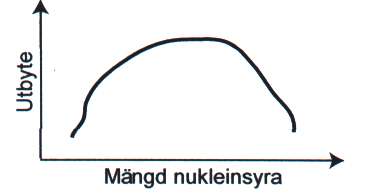
Utbytet beroer också på mängden nukleinsyra i provet från början (figur 7). Har man mycket stor mängd nukleinsyra kan matrisen mättas, vilket ger ett lågt utbyte. Har man mycket liten mängd blir det svårt att eluera. Genom att tillsätta en inert bärare som binder in till matrisen, t ex tRNA eller glykogen, kan man öka utbytet av den eftersökta nukleinsyran i extraktet.
Figur 7. Utbyte av nukleinsyra i förhållande till nukleinsyramängden i provmaterialet. Bild: Håkan Janson
Automatiserad nukleinsyraextraktion[redigera]
Det finns flera leverantörer av robotar för nukleinsyraextraktion. De flesta utnyttjar silika-täckta paramagnetiska kulor som har en stor fördel gentemot silika-kolonner i det att kulorna ger en stor yta i förhållande till volym. Inbindning, tvätt och eluering blir effektivare då den utförs i suspension, samtidigt som det går åt mindre volymer av lösningar för att nå samma reningsgrad. Vid byte av buffertar samlar en magnet ihop kulorna så att den använda lösningen lätt kan tas bort och ersättas med nästa lösning. I princip alla nukleinsyraextraktorer för mikrobiologisk användning bygger på Boom-extraktion. Provet lyseras med det starkt denaturerande ämnet guanidin-isothiocyanat. Den stabiliserade nukleinsyran binder till en silika-matris och tvättas med en guanidin-isotiocyanat-innehållande buffert med hög jonstyrka. Eftersom nukleaser har avlägsnats i föregående tvätt kan nukleinsyran nu tvättas vidare med etanol och aceton och sedan torkas innan den elueras i en buffert med låg jonstyrka. Till extraktorerna kan man använda kommersiella kit anpassade för renfarmställning av nukleinsyror från olika provtyper.
Val av extraktionskit[redigera]
Vilket extraktionskit man väljer är avhängigt av vilken typ av nukleinsyra man önskar påvisa eller analysera, hur hög reningsgrad som önskas, vilken mängd som behövs samt vilket provmaterial man utgår ifrån. Nedan listas några exempel på extraktionskit som finns att tillgå, både för automatiserade och manuella system.
- DNA minipreparation från helblod (ger en liten mängd från en liten volym)
- DNA midiprepation från helblod (ger en större mängd från en större volym)
- DNA minipreparation från vävnad, bakterier och svampar (kraftigare lyseringssteg)
- Virus RNA från plasma och serum
- Virus total nukleinsyra från plasma och serum
- mRNA från celler
- Total RNA från celler
- Total RNA från vävnad (kraftigare lyseringssteg)
Vissa kit kan användas till flera applikationer genom att förbehandling och extraktionsbetingelser varieras. För vävnadsprov kan provet förbehandlas genom homogenisering och proteinas K-behandling innan det används för extraktion med ett vanligt DNA-kit. Beroende på provmaterial kan provvolymer och volym på tvätt- respektive elueringslösningar samt inkubationstider varieras utifrån hur komplext provmaterialet är.
En bekväm lösning för ett mikrobiologiskt laboratorium är att använda ett kit som renar fram total nukleinsyra från provmaterial så att man kan analysera både DNA- och RNA-innehållande agens i samma extrakt. Ett sådant kit lämpar sig bäst för prov med låga halter av nukleinsyra, t ex serum, plasma och eventuellt likvor och urin, medan det inte är lämpligt att använda för RNA-extraktion av cellrika provmaterial som helblod och vävnader. Humanblod har mellan 4 och 10 miljoner leukocyter per mL. I varje leukocyt finns ca 6 pg DNA vilket gör att varje mL helblod innehåller mellan 25 - 60 μg DNA. Mängden RNA är betydligt lägre, mellan 1 till 5 μg per mL. Om man är ute efter RNA i ett prov som huvudsakligen består av DNA, så finns det risk att man mättar matrisen med DNA som därmed konkurrerar ut RNA. Därför brukar kit för RNA preparation från cellrika provmaterial innehålla DNaser som hydrolyserar DNA. I vissa fall innehåller kit för DNA-rening RNaser, men här är det snarast en fråga om att få en så ren DNA-produkt som möjligt för att minimera interferens av RNA i genetiska analyser etc.
På senare år har särskilda extraktionskit anpassade för användning inom bakteriologisk och mykologisk molekylärbiologi börjat att marknadsföras. Dessa kit tillverkas så att förekomsten av bakterie- och svamp-DNA i reagenserna minimerats, vilket innebär att kiten kan användas för extraktion av prov som ska analyseras med breda amplifieringsmetoder såsom rRNA-genanalys. De lämpar sig även väl för Legionella-diagnostik då man ibland hittat spår av Legionella-DNA i vanliga kiseldioxidmatriser. Man bör eftersträva att genomgående använda garanterat mikrobfria reagenser för all molekylär mikrobiologi.
Det finns även reagenser som anrikar de agens man vill extrahera. Inom livsmedelsindustrin använder man ofta specifika antikroppar, bundna till paramagnetiska kulor, för att anrika mikrober från utspädda lösningar. Det finns även speciella silika-täckta paramagnetiska kulor som har affinitet för bakterier vilka kan användas både för anrikning och för extraktion av provmaterial.
Nukleinsyrakvalitet[redigera]
Parametrar som används för att beskriva en extraktionsmetods effektivitet är utbyte, renhet, storlek, fragmentering och PCR-inhibition.
Utbytet ska naturligtvis vara så högt som möjligt utan att ge ett för utspätt extrakt. Mängden nukleinsyra i extraktet kan beräknas genom spektrofotometrisk mätning vid 260 nm eller genom fluorometrisk bestämning efter tillsats av pico-green eller motsvarande färgämne.
Renheten av DNA bedöms ofta som en kvot mellan extraktets absorbans vid 260 nm (där DNA absorberar bäst) och 280 nm (där proteiner och fenol absorberar bäst). För rent DNA ska A260/A280 kvoten vara cirka 1,8. För RNA räcker det inte att mäta A260/A280 kvoten (som ska vara cirka 2), utan man mäter också kvoten mellan A260/A230 som bör vara över 1 för en ren preparation. Det bästa är att ta ett fullt UV-spektrum, dvs mäta absorbansen över hela området mellan A230 och A260, för att mäta renheten hos RNA-extraktionen. Man kan också uppskatta renhet genom att analysera extraktet med gelelektrofores. Det är också möjligt att uppskatta mängden nukleinsyra i relation till kända mängder med gelelektrofores eller med realtids-PCR. För att koncentrationsbestämning med realtids-PCR ska bli korrekt krävs att provet inte innehåller några PCR-inhibitorer.
Storleken och fragmenteringsgraden av extraherat material uppskattas efter gelelektrofores. Extraherat DNA bör vara så stort som möjligt och inte vara fragmenterat. Fragmenteringsgrad kan också uppskattas genom amplifieringsbarhet, till exempel i extrakt från paraffininbäddad vävnad, så länge man visat att extraktet inte innehåller PCR-inhibitorer.
Ett av huvudskälen till att extrahera provmaterial inför en PCR-analys är att avlägsna PCR-inhibitorer som hämmar reaktionen och därmed riskerar att göra positiva prov falskt negativa. Eventuell förekomst av inhibitorer påvisas lättast genom tillsatts av en känd mängd kontrollnukleinsyra till det extrakt man vill undersöka. Om PCR-signalen från kontrollen blir den förväntade finns det inga inhibitorer i provet.
Andra faktorer att tänka på, när man väljer extraktionsmetod, är att proceduren skall vara säker ur kontaminationssynpunkt. Man måste undvika överföring av nukleinsyra mellan olika prov. Även stabiliteten av extraherad nukleinsyra är av intresse, speciellt om man planerar att förvara provet en längre tid.
Kvalitetssäkring[redigera]
Oavsett vilken extraktionsmetod man väljer är det viktigt att man har utvärderat att metoden utför det som den är avsedd för. Det finns kommersiella extraktionsmetoder som är godkända för in-vitro diagnostik (IVD-godkända). För dessa metoder gäller rigorösa begränsningar för vilken typ av agens respektive provmaterial de får användas till. Anledningen till detta är att metoder, som man exempelvis optimerat för extraktion av RNA-virus i likvor, förmodligen inte lämpar sig för att extrahera samma agens i feces. Detsamma får antas gälla beträffande användning av olika extraktionskit för samma agens och provtyp.
REFERENSER[redigera]
- 1. Chomczynski,P. and Sacchi,N. Anal Biochem. 1987; 162: 156-159.
- 2. Boom R, Sol CJ, Salimans MM, Jansen CL, Wertheim-van Dillen PM, van der Noordaa J J Clin Microbiol 1990; 28:495–503