Bilaga 1: Realtids-PCR för påvisande av plasmidburen AmpC (ESBLM)
Artikeln publicerad mars 2012.
Till innehållsförteckningen Referensmetodik:Smittskyddslagens sjukdomar
Till artikeln Screeningmetodik för ESBL
Bilaga 1: Realtids-PCR för påvisande av plasmidburen AmpC (ESBLM)[redigera]
Inledning[redigera]
PCR utgör referensmetod för påvisande av plasmidburen AmpC (ESBLM). Den nedan beskrivna metoden är avsedd att vara ett exempel bland andra tänkbara på modifierad variant av en multiplex PCR som i orginalutförande beskrevs av Perez-Perez (Ref 1), senare modifierad av Brolund et al (Ref 2). Den ursprungliga metoden beskrev påvisandet av sex subgrupper (CIT, MOX, FOX, DHA, ACC och EBC) av plasmidmedierad AmpC (pAmpC, ESBLM), men eftersom hittills två subgrupper (CIT och enstaka DHA) dominerar i Sverige, beskrivs nedan en metod för påvisande av dessa. Metod för påvisande av övriga subgrupper kan erhållas från Folkhälsomyndigheten. Det finns i dag även kommersiella kit (t ex från Check-Points) för påvisande av plasmidburen AmpC.
PCR-metod[redigera]
Nedanstående metodbeskrivning är avsedd som exempel på PCR-påvisning av plasmidburen AmpC.
- Provmaterial: Utväxta kolonier av bakterier tillhörande Enterobacteriaceae med fenotypisk misstanke om ESBLM kan användas som utgångsmaterial.
- Instrument: LightCycler 2.1 Roche
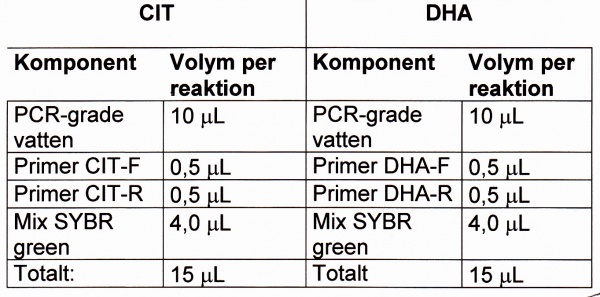
- Reagens: LightCycler Fast start DNA MasterPLUS SYBR Green 1 och H2O PCR-grade (båda Roche Diagnostics Scandinavia AB).
- Primerpar:
- CIT: amplimer har storleken 462 bp
- DHA: amplimer har storleken 218 bp
- Primers 10 μM:
- CIT-F 5'-TGG CCA GAA CTG ACA GGC AAA-3'
- CIT-R 5’-TTT CTC CTG AAC GTG GCT GGC-3’
- DHA-F 5’-AAC TTT CAC AGG TGT GCT GGG T-3’
- DHA-R 5’-GCT GCC ACT GCT GAT AGA A-3’
- Referensstammar:
- E. coli CCUG 58543 (CIT)
- Klebsiella pneumoniae CCUG 58545 (DHA)
Metod-kontroll[redigera]
Vid varje analystillfälle testas förutom patientprov även (positiva kontroller) kontrollstammar CCUG 58543 (CIT), CCUG 58545 (DHA) och (negativa kontroller) som blankprov H2O PCR-grade (PCR-rent vatten samt NTC (PCR-mix utan templat).
Utförande[redigera]
Moment 1 (DNA-preparation):
- Märk upp sterila Eppendorfrör 1,5 mL. Ett rör för varje misstänkt isolat, ett för var kontrollstam och ett för vatten. Tillsätt 100 mL H2O PCR-grade till varje rör.
- Lös upp 1 CFU i röret. Inkubera proven, 98ºC, 15 minuter i värmeblock.
- Centrifugera 13 000 rpm i 5 minuter. Supernatanten används som templat.
Moment 2 (Tillverkning av PCR-mix):
- Master Mix SYBR green 1: Beredning enligt gängse metod.
- PCR-mix: Blanda en mix per komponent: antal rör + en NTC + en extra.
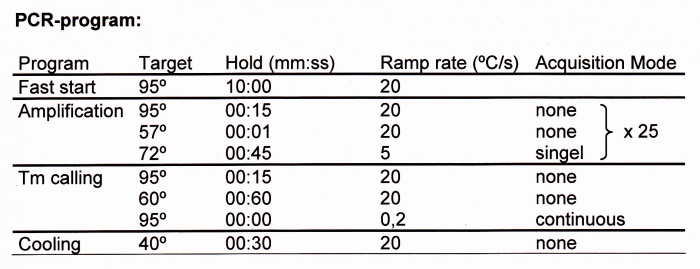
Moment 3 (Provsättning och start av LightCycler):
Läs av amplifierings- och smältpunkts-kurvorna
Bedömning[redigera]
- 1. Kontrollera att kontrollerna slår som förväntat; CCUG 58543 (CIT) pos i CIT-mix och CCUG 58545 (DHA) positiv i DHA-mix.
- 2. Tm CIT bör ligga 90 +/- 1ºC, Tm DHA bör ligga 91 +/- 1ºC.
- 3. Tm kontroll-Tm prov ska vara +/- 1,00ºC för att säkerställa specificitet av PCR-produkt.
För verifierad genotypisk ESBLM krävs alltså:
- A) Fenotypiska karaktäristika
- B) Tm +/- 1,00ºC från positiva kontrollen
Positivt utfall vid smältpunktanalys tolkas som uttryck av CIT eller DHA.
Negativt utfall vid smältpunktsanalys tolkas som kromosomalt medierad AmpC.
REFERENSER[redigera]
- 1. Perez-Perez FJ, Hanson ND. Detection of plasmid-mediated AmpC beta-lactamase genes in clinical isolates by using multiplex PCR. J Clin Microbiol. 2002 Jun;40(6):2153-62.
- 2. Brolund A, Wisell KT, Edquist PJ, Elfstrom L, Walder M, Giske CG. Development of a real-time SYBRGreen PCR assay for rapid detection of acquired AmpC in Enterobacteriaceae. J Microbiol Methods. 2010 Sep;82(3):229-33.