Skillnad mellan versioner av "Sekvensering"
Hoppa till navigering
Hoppa till sök
| Rad 10: | Rad 10: | ||
*8. Kulor med enkelsträngat DNA som har amplifierats från den magnetbundna primern anrikas. De tvättade kulorna med DNA blandas med en lösning med större polystyrenkulor vilka har en linker på ytan som kan binda B-adaptorn från DNA-kulan. Denna ”kulmix” läggs ovanpå en glycerollösning och centrifugeras. Efter centrifugering hamnar kulor utan DNA i en pellet och kulor som är bundna till polystyren kulan ligger kvar i en fas ovanpå glycerolen. Dessa kulor tvättas och DNA denatureras så det släpper från polystyrenkulan. | *8. Kulor med enkelsträngat DNA som har amplifierats från den magnetbundna primern anrikas. De tvättade kulorna med DNA blandas med en lösning med större polystyrenkulor vilka har en linker på ytan som kan binda B-adaptorn från DNA-kulan. Denna ”kulmix” läggs ovanpå en glycerollösning och centrifugeras. Efter centrifugering hamnar kulor utan DNA i en pellet och kulor som är bundna till polystyren kulan ligger kvar i en fas ovanpå glycerolen. Dessa kulor tvättas och DNA denatureras så det släpper från polystyrenkulan. | ||
*9. 3’ ändan av dessa strängar modifieras med terminal transferas så att de binder kovalent till glas. Kulorna deponeras på ett glas som kan delas in i ett, fyra eller åtta fält. | *9. 3’ ändan av dessa strängar modifieras med terminal transferas så att de binder kovalent till glas. Kulorna deponeras på ett glas som kan delas in i ett, fyra eller åtta fält. | ||
| − | *10. DNA sekvenseras med ligassekvensering. Ofta används 5 olika primers som binder olika långt ut från adaptorn. Proben som ligeras till primern har färgmolekylen på sig. Proben är en oktamer med enbart två unika baser, 256 olika prober per färg, totalt 1024 olika prober finns i probemixen ( | + | *10. DNA sekvenseras med ligassekvensering. Ofta används 5 olika primers som binder olika långt ut från adaptorn. Proben som ligeras till primern har färgmolekylen på sig. Proben är en oktamer med enbart två unika baser, 256 olika prober per färg, totalt 1024 olika prober finns i probemixen (4<math>^5</math>). När proben bundit läses färgen av från varje kula i fyra olika kanaler. Efter detta klyvs 5’ änden bort på proben så nästa probe kan ligeras. När signal från sista proben detekterats rensas templatet och nästa primer kan binda in ett steg före föregående primers bindningsställe. Vid sekvensering av fragmentbibliotek läses 50 baser med start från adaptor. Fragmentet kan vara av olika längd men enbart 25-50 baser sekvenseras. Under våren 2009 kommer en uppgradering till SOLiD med möjlighet till läsning av 75 baser. |
| − | *11. Att sekvensera ett fragmentbibliotek tar idag cirka 3-5 dagar. Man kan då erhålla cirka 100* | + | *11. Att sekvensera ett fragmentbibliotek tar idag cirka 3-5 dagar. Man kan då erhålla cirka 100*<math>10^9</math> baser vid en sekvenseringsomgång. Idag tar hanteringen av all sekvenseringsdata längst tid. |
| + | |||
| + | |||
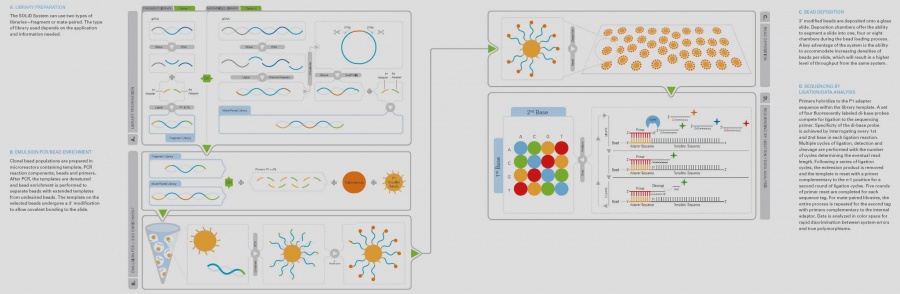
[[Fil:Cms 053243.jpg|thumb|center|900px|||'''Figur 24. '''. Sekvensering enligt SOLiD-teknik. Texten i figuren beskriver de olika stegen. ''Bild: Applied Biosystems en del av Life Technologies Group'', publicerad med företagets godkännande. | [[Fil:Cms 053243.jpg|thumb|center|900px|||'''Figur 24. '''. Sekvensering enligt SOLiD-teknik. Texten i figuren beskriver de olika stegen. ''Bild: Applied Biosystems en del av Life Technologies Group'', publicerad med företagets godkännande. | ||
'''FÖR LÄSBAR BILD, KLICKA PÅ BILDEN, DU KOMMER DÅ TILL FILSIDAN DÄR DU ÅTER KAN KLICKA PÅ BILDEN TILL LÄSBARHET''']] | '''FÖR LÄSBAR BILD, KLICKA PÅ BILDEN, DU KOMMER DÅ TILL FILSIDAN DÄR DU ÅTER KAN KLICKA PÅ BILDEN TILL LÄSBARHET''']] | ||
| + | |||
== REFERENSER == | == REFERENSER == | ||
Versionen från 4 september 2010 kl. 07.23
SOLiD
- 1. Extraktion av DNA/RNA som skall sekvenseras.
- 2. Mekanisk klyvning (sonikering) av DNA/cDNA.
- 3. Separation av fragmenten via gelelektrofores. Fragment motsvarande en bestämd längd (ca 100 bp vid ett fragmentbibliotek och längre fragment vid t.ex genomisk kartläggning) skärs ut. Rening av fragmenten från gelen.
- 4. Korta DNA-strängar (adaptrar) A och B ligeras till båda ändarna (5’ och 3’) på templat DNA.
- 5. PCR-amplifiering av templatet med hjälp av primers riktade mot adapterregionerna. Genom att begränsa primerkoncentrationen för A-regionen och tillsätta magnetiska kulor som har den primern får man amplifiering av DNA från primer kopplade till magnetkulorna. Man försöker uppnå ett 1:1 förhållande mellan kulor och templatet.
- 6. PCR-amplifieringen sker i en vatten-oljeemulsion.
- 7. Emulsionen bryts när PCRen är färdig genom att butanol tillsätts och oljan tvättas bort.
- 8. Kulor med enkelsträngat DNA som har amplifierats från den magnetbundna primern anrikas. De tvättade kulorna med DNA blandas med en lösning med större polystyrenkulor vilka har en linker på ytan som kan binda B-adaptorn från DNA-kulan. Denna ”kulmix” läggs ovanpå en glycerollösning och centrifugeras. Efter centrifugering hamnar kulor utan DNA i en pellet och kulor som är bundna till polystyren kulan ligger kvar i en fas ovanpå glycerolen. Dessa kulor tvättas och DNA denatureras så det släpper från polystyrenkulan.
- 9. 3’ ändan av dessa strängar modifieras med terminal transferas så att de binder kovalent till glas. Kulorna deponeras på ett glas som kan delas in i ett, fyra eller åtta fält.
- 10. DNA sekvenseras med ligassekvensering. Ofta används 5 olika primers som binder olika långt ut från adaptorn. Proben som ligeras till primern har färgmolekylen på sig. Proben är en oktamer med enbart två unika baser, 256 olika prober per färg, totalt 1024 olika prober finns i probemixen (4). När proben bundit läses färgen av från varje kula i fyra olika kanaler. Efter detta klyvs 5’ änden bort på proben så nästa probe kan ligeras. När signal från sista proben detekterats rensas templatet och nästa primer kan binda in ett steg före föregående primers bindningsställe. Vid sekvensering av fragmentbibliotek läses 50 baser med start från adaptor. Fragmentet kan vara av olika längd men enbart 25-50 baser sekvenseras. Under våren 2009 kommer en uppgradering till SOLiD med möjlighet till läsning av 75 baser.
- 11. Att sekvensera ett fragmentbibliotek tar idag cirka 3-5 dagar. Man kan då erhålla cirka 100* baser vid en sekvenseringsomgång. Idag tar hanteringen av all sekvenseringsdata längst tid.
REFERENSER
- 1 Maxam AM, Gilbert W. A new method for sequencing DNA. Proc. Natl. Acad. Sci. U.S.A. 1977;74:560-4.
- 2 Sanger F, Nicklen S, Coulson AR. DNA sequencing with chain-terminating inhibitors. Proceedings of the National Academy of Sciences USA 1977;74: 5463-5467.
- 3 Nyren P. Enzymatic method for continuous monitoring of DNA polymerase activity. Anal. Biochem. 1987;167:235-8.
- 4 Ahmadian A, Ehn M, Hober S. 2006. Pyrosequencing: History, biochemistry and future. Clinica. Chimica. Acta. 2006;363(1-2): 83-94.
- 5 Shendure JA, Porreca GJ, Church GM. Overview of DNA sequencing strategies. Current Protocols in Mol. Biol. 2008; 7.1.1-11.
- 6 Ausubel FM, Albright LM, Ju J. DNA sequencing. Current Protocols in Mol. Biol. 2008; 7.0.1-15.
- 7 Metzker ML. Emerging technologies in DNA sequencing. Genome Res. 2005;15(12):1767-76.
- 8 Ziebolz B, Droege M. Toward a new era in sequencing. Biotechnol Annu Rev. 2007;13:1-26.
- 9 Bentley DR. Whole-genome re-sequencing. Curr Opin Genet Dev. 2006;16(6):545-52.
- 10 Venter JC, Smith HO, Hood L. A new strategy for genome sequencing. Nature 1996; 381, 364-6.