Skillnad mellan versioner av "Bilaga 8 (CNS)"
(Skapade sidan med '''Till innehållsförteckningen för Referensmetodik: Infektioner i centrala nervsystemet'' ---- == Bilaga 8 == === Cytomegalovirus. Detektion av CMV DNA med PCR-tekn;k en…') |
|||
| (2 mellanliggande sidversioner av samma användare visas inte) | |||
| Rad 21: | Rad 21: | ||
PCR-apparat (TC- 1, Perkin Elmer) | PCR-apparat (TC- 1, Perkin Elmer) | ||
| − | + | Variabla pipetter, fördelade på arbetsstationer | |
Elektroforesutrustning | Elektroforesutrustning | ||
| Rad 77: | Rad 77: | ||
''2:a amplifieringen'' | ''2:a amplifieringen'' | ||
| − | *CMV3 5’-GCC GAT CCT CTG AGA GTC TGC TCT C- | + | *CMV3 5’-GCC GAT CCT CTG AGA GTC TGC TCT C-3’ |
| − | *CMV4 | + | *CMV4 5’-CAG CCA CAA TI’A CTG AGG ACA GAG-3’ |
Storlek på PCR-fragment: 191 baspar | Storlek på PCR-fragment: 191 baspar | ||
| Rad 88: | Rad 88: | ||
| − | ''' | + | '''MgCl<sub>2</sub>,''' Perkin Elmer |
Koncentration: 15 mmol/L, | Koncentration: 15 mmol/L, | ||
| Rad 104: | Rad 104: | ||
'''Enzym,''' Perkin Elmer | '''Enzym,''' Perkin Elmer | ||
| − | Taq polymeras 5 U/ | + | Taq polymeras 5 U/μL |
Förvaras vid -20 °C. | Förvaras vid -20 °C. | ||
| Rad 116: | Rad 116: | ||
'''10x TBE-buffert,''' Karolinska sjukhuset (KS) | '''10x TBE-buffert,''' Karolinska sjukhuset (KS) | ||
| − | pH 8,0-8,4. Späds i | + | pH 8,0-8,4. Späds i H<sub>2</sub>O till lx TBE-buffert. |
Förvaras i rumstemperatur. | Förvaras i rumstemperatur. | ||
| Rad 135: | Rad 135: | ||
| − | *[[Fil: | + | *[[Fil:CNStabellbil8b.jpg]] |
'''Provberedning inför 1:a amplifieringen''' | '''Provberedning inför 1:a amplifieringen''' | ||
| − | * Frys likvor vid <-18 °C. | + | * Frys likvor vid <=-18 °C. |
* Tina i rumstemperatur. | * Tina i rumstemperatur. | ||
* Sätt 10 μL likvor till 0,5 mL PCR-rör i duplikat. | * Sätt 10 μL likvor till 0,5 mL PCR-rör i duplikat. | ||
| − | * Tillsätt 1 droppe ( | + | * Tillsätt 1 droppe (cirka 25 μL) mineralolja. Stäng locket ordentligt. |
* Hetta upp likvorprovt i PCR-rören med olja i PCR-maskinen 95 °Ci—10 min. | * Hetta upp likvorprovt i PCR-rören med olja i PCR-maskinen 95 °Ci—10 min. | ||
* De olika momenten i proceduren utförs på fyra separata arbetsstationer för att minimera riskerna för kontamination av prov-DNA med amplifierad produkt. | * De olika momenten i proceduren utförs på fyra separata arbetsstationer för att minimera riskerna för kontamination av prov-DNA med amplifierad produkt. | ||
| Rad 151: | Rad 151: | ||
*Sätt upp PCR-rör i still (sättningsbox) inklusive de förbehandlade , provrören enligt analysprotokoll. | *Sätt upp PCR-rör i still (sättningsbox) inklusive de förbehandlade , provrören enligt analysprotokoll. | ||
*Stäng rören och märk locken med nummer enligt analysprotokoll Oppna ett rör i taget i nummerordning. | *Stäng rören och märk locken med nummer enligt analysprotokoll Oppna ett rör i taget i nummerordning. | ||
| − | * Tillsätt 10 μL | + | * Tillsätt 10 μL H<sub>2</sub>0 till vattenkonltroll och en droppe olja ovanpå |
* Tillsätt 40 μL mastermix/rör under oljan. | * Tillsätt 40 μL mastermix/rör under oljan. | ||
* Stäng rören ordentligt. | * Stäng rören ordentligt. | ||
| Rad 170: | Rad 170: | ||
| − | |||
| − | |||
| − | * Gör | + | '''Provberedning inför 2:a amplifieringen''' |
| + | |||
| + | * Gör Mastermix 2 med inre primers (ren plats). | ||
* Sätt upp PCR-rör i ställ. | * Sätt upp PCR-rör i ställ. | ||
* Fördela 47,5 μL mastermix/rör | * Fördela 47,5 μL mastermix/rör | ||
| Rad 199: | Rad 199: | ||
'''Elfores''' | '''Elfores''' | ||
| − | * Gör i ordning 1,5 %-ig agaros med 0,5 μg EtBr/mlL agaros i lx | + | * Gör i ordning 1,5 %-ig agaros med 0,5 μg EtBr/mlL agaros i lx TBE-buffert. Gjut en gelform med kammar. Lägg ner den stela gelen i elektroforesbadet med 0,5 μg EtBr/mL TBEbuffert. |
| − | * På en remsa | + | * På en remsa parafilm droppas 2 μL 6x loadingbUffe1 och blandas med 10 μL PCRprodukt. Sätt 10 μL produkt/hål i gelen. |
* Starta aggregatet (140 V). | * Starta aggregatet (140 V). | ||
* Låt gelen gå i ca 20-30 min. | * Låt gelen gå i ca 20-30 min. | ||
| − | * Stäng av aggregatet och ta upp gelen. Fotografera den på UV-bordet Ta ett kort till analysprotokoflet | + | * Stäng av aggregatet och ta upp gelen. Fotografera den på UV-bordet. |
| + | * Ta ett kort till analysprotokoflet | ||
| Rad 209: | Rad 210: | ||
* Analysen godkänns om 5 och/eller 10 fg av renat CMV DNA kan detekteras. | * Analysen godkänns om 5 och/eller 10 fg av renat CMV DNA kan detekteras. | ||
* Kontrollera storleken av amplifikat jämfört med den positiva kontrollen. | * Kontrollera storleken av amplifikat jämfört med den positiva kontrollen. | ||
| − | * Om bägge portionerna av likvorprovet blir positiva indikerar fyndet en CMV- | + | * Om bägge portionerna av likvorprovet blir positiva indikerar fyndet en CMV-orsakad CNS-infektion |
* Om endast ena portionen blir positiv sätts provet om. Om fyndet kan repeteras svaras provet ut positivt, annars negativt. | * Om endast ena portionen blir positiv sätts provet om. Om fyndet kan repeteras svaras provet ut positivt, annars negativt. | ||
* Om oklarheter: Sätt om provet, ev i spädning eller efter DNA-extration om det ger smear. | * Om oklarheter: Sätt om provet, ev i spädning eller efter DNA-extration om det ger smear. | ||
Nuvarande version från 19 december 2009 kl. 13.28
Till innehållsförteckningen för Referensmetodik: Infektioner i centrala nervsystemet
Bilaga 8[redigera]
Cytomegalovirus. Detektion av CMV DNA med PCR-tekn;k enligt metod från Sahlgrenska sjukhuset, Göteborg, modifierad vid Smittskyddsinstitutet[redigera]
Provtagningsmaterial
Cerebrospinalvätska (Csv)
Förvaras vid 4 °C eller -20 °C fram till transporten
Transport vid rumstemperatur
Apparatur
PCR-apparat (TC- 1, Perkin Elmer)
Variabla pipetter, fördelade på arbetsstationer
Elektroforesutrustning
Polaroidkamera
UV-ljusbord
Förbrukningsmaterial
0,5 mL PCR-rör
1,5 mL Eppendorfrör
0,5-10 μL tippar utan filter
0,5-10 μL tippar med filter
10-200 μL tippar utan filter
100-1000 μL tippar utan filter
Reagens
H20: Sterilt vatten (dest.vatten) av hög renhetsgrad
Olja: Mineralolja, light
Primers: Pharmacia Biotech
Förvaras i -20 °C i Rena rummet
Koncentrationer:
1,4 μmol/L: CMVI och CMV2
2,8 μmol/L: CMV3 och CMV4
Samtliga primers är lokaliserade inom fjärde exonet av Major Immediate early gene (UL 122-123)
1:a amplifieringen
- CMV1 5’-GCG CCG CAT TGA GGA GAT CTG CAT-3’
- CMV2 5’-GAG CAC CCT CCT CCT crr CCT CAT-3’
Storlek på PCR-fragment: 299 baspar
2:a amplifieringen
- CMV3 5’-GCC GAT CCT CTG AGA GTC TGC TCT C-3’
- CMV4 5’-CAG CCA CAA TI’A CTG AGG ACA GAG-3’
Storlek på PCR-fragment: 191 baspar
10 x PCR buffert II, Perkin Elmer
Förvaras vid 4 °C.
MgCl2, Perkin Elmer
Koncentration: 15 mmol/L,
Förvaras vid 4 °C.
dNTP, Pharmacia Biotech
Koncentration: 2 mmol/L används i en blandning av samtliga nukleotider.
Förvaras vid -20 °C.
Enzym, Perkin Elmer
Taq polymeras 5 U/μL
Förvaras vid -20 °C.
Agaros NA, Pharmacia Biotech
Förvaras som pulver i rumstemperatur.
10x TBE-buffert, Karolinska sjukhuset (KS)
pH 8,0-8,4. Späds i H2O till lx TBE-buffert.
Förvaras i rumstemperatur.
Etidiumbromid (EtBr) KS
Koncentration: 10 mg/mL.
Förvaras i rumstemperatur.
6x Loading buffert, Scandinavian Diagnostic Services (SDS)
Blue orange loading buffert.
Förvaras vid 4 °C.
Provberedning inför 1:a amplifieringen
- Frys likvor vid <=-18 °C.
- Tina i rumstemperatur.
- Sätt 10 μL likvor till 0,5 mL PCR-rör i duplikat.
- Tillsätt 1 droppe (cirka 25 μL) mineralolja. Stäng locket ordentligt.
- Hetta upp likvorprovt i PCR-rören med olja i PCR-maskinen 95 °Ci—10 min.
- De olika momenten i proceduren utförs på fyra separata arbetsstationer för att minimera riskerna för kontamination av prov-DNA med amplifierad produkt.
- Skriv analysprotokoll där 1:a röret är en vattenkontroll och patientprov analyseras i duplikat. Vattenkontroller sätts mellan olika patientduplikat. Positiva kontroller sättes sist.
- Förbehandla likvor enil. provbehandling ovan (sättningsbox).
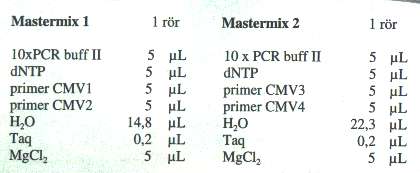
- Plocka fram reagenserna (ren plats) och blanda mastermix 1 i Eppendorfrör 1,5 mL.
- Sätt upp PCR-rör i still (sättningsbox) inklusive de förbehandlade , provrören enligt analysprotokoll.
- Stäng rören och märk locken med nummer enligt analysprotokoll Oppna ett rör i taget i nummerordning.
- Tillsätt 10 μL H20 till vattenkonltroll och en droppe olja ovanpå
- Tillsätt 40 μL mastermix/rör under oljan.
- Stäng rören ordentligt.
- Sätt de positiva kontrollerna sist. Eventuella uppspädning& av positiva kontroller görs efter det att proven är satta och locken ordentligt tillsiutna.
1:a amp1ifieringen
Kör följande program:
5 min 92 °C
(94 °C 30 sek, 57 °C 30 sek, 72 °C I min)x20 cykler
1 min72°C
sök 4 °C
Provberedning inför 2:a amplifieringen
- Gör Mastermix 2 med inre primers (ren plats).
- Sätt upp PCR-rör i ställ.
- Fördela 47,5 μL mastermix/rör
- Tillsätt 1 droppe olja ovanpå.
- Stäng locken och märk locken med nummer enl. analysprotokoll.
- Tag med rören till nestningsplats.
- För över 2,5 μL prov från 1:a steget till de nya rören.
Stor risk för kontamination, öppna därför ett rör i taget.
2:a amplifieringen
Kör följande program:
5 min 92 °C
(94 °C 30 sek, 57 °C 30 sek, 72 °C 30Sek)X30 cykler
1 min 72 °C
Sök 4 °C
Elfores
- Gör i ordning 1,5 %-ig agaros med 0,5 μg EtBr/mlL agaros i lx TBE-buffert. Gjut en gelform med kammar. Lägg ner den stela gelen i elektroforesbadet med 0,5 μg EtBr/mL TBEbuffert.
- På en remsa parafilm droppas 2 μL 6x loadingbUffe1 och blandas med 10 μL PCRprodukt. Sätt 10 μL produkt/hål i gelen.
- Starta aggregatet (140 V).
- Låt gelen gå i ca 20-30 min.
- Stäng av aggregatet och ta upp gelen. Fotografera den på UV-bordet.
- Ta ett kort till analysprotokoflet
Bedömning
- Analysen godkänns om 5 och/eller 10 fg av renat CMV DNA kan detekteras.
- Kontrollera storleken av amplifikat jämfört med den positiva kontrollen.
- Om bägge portionerna av likvorprovet blir positiva indikerar fyndet en CMV-orsakad CNS-infektion
- Om endast ena portionen blir positiv sätts provet om. Om fyndet kan repeteras svaras provet ut positivt, annars negativt.
- Om oklarheter: Sätt om provet, ev i spädning eller efter DNA-extration om det ger smear.