Skillnad mellan versioner av "Realtids-PCR"
| (7 mellanliggande sidversioner av samma användare visas inte) | |||
| Rad 21: | Rad 21: | ||
Den vanligaste typen av hydrolysprobe är ''TaqMan''-probe. Den är i sin 5’-ände inmärkt med en fluorofor, en så kallad reporter och i sin 3’-ände inmärkt med en quencher. Vid amplifiering binder proben in till mål-DNA och under elongeringsfasen hydrolyseras proben, bryts ned, av Taq-polymerasets 5’-exonukleas aktivitet varvid reporter och quencher skiljs åt ('''figur 9'''). Energin från den fria och exciterade reportern absorberas inte längre av quenchern utan tas nu upp av detektorn i realtids-PCR-instrumentet. Specificiteten för en TaqMan-probe kan ökas genom att man i 3’-änden fäster en så kallad ''minorgroove binder'' (MGB)-molekyl. PCR-reaktionen blir nu mer robust då MGB-molekylen binder hårdare till DNA-molekylen, vilket innebär att probens smältvärde (Tm) ökar. Hydrolysprober fungerar bäst vid tvåstegs-PCR, vilket innebär att annealing och elongeringsfaserna slås samman till en fas i PCR-reaktionen. | Den vanligaste typen av hydrolysprobe är ''TaqMan''-probe. Den är i sin 5’-ände inmärkt med en fluorofor, en så kallad reporter och i sin 3’-ände inmärkt med en quencher. Vid amplifiering binder proben in till mål-DNA och under elongeringsfasen hydrolyseras proben, bryts ned, av Taq-polymerasets 5’-exonukleas aktivitet varvid reporter och quencher skiljs åt ('''figur 9'''). Energin från den fria och exciterade reportern absorberas inte längre av quenchern utan tas nu upp av detektorn i realtids-PCR-instrumentet. Specificiteten för en TaqMan-probe kan ökas genom att man i 3’-änden fäster en så kallad ''minorgroove binder'' (MGB)-molekyl. PCR-reaktionen blir nu mer robust då MGB-molekylen binder hårdare till DNA-molekylen, vilket innebär att probens smältvärde (Tm) ökar. Hydrolysprober fungerar bäst vid tvåstegs-PCR, vilket innebär att annealing och elongeringsfaserna slås samman till en fas i PCR-reaktionen. | ||
| − | [[Fil:molekylfig9.jpg | + | [[Fil:molekylfig9.jpg|thumb|center|500px|||'''Figur 9'''. TaqMan-probe. (A) Principen för en TaqMan-probe är att probe och primer binder in till templatet där en intakt probe inte avger någon emissionsenergi. (B) Vid elongering avlägsnas fluoroforen (reporter) vilket medför att man kan mäta energin från den fria fluoroforen. ''Bild: © QIAGEN, all rights reserved'']] |
| − | |||
| − | '''Figur 9'''. TaqMan-probe. (A) Principen för en TaqMan-probe är att probe och primer binder in till templatet där en intakt probe inte avger någon emissionsenergi. (B) Vid elongering avlägsnas fluoroforen (reporter) vilket medför att man kan mäta energin från den fria fluoroforen. ''Bild: © QIAGEN, all rights reserved'' | ||
=== FRET-probe === | === FRET-probe === | ||
| Rad 29: | Rad 27: | ||
Första exemplet på en hybridiseringsprobe är ''Fluorescence Resonance Energy Transfer''-probe som förkortas ''FRET''-probe. Den består av två oligonukleotider inmärkta med varsin fluorofor men med olika excitations- och emissionsspektra. Proberna är designade så att de hybridiserar, binder in, till mål-DNA med endast några basers mellanrum. Den första probens fluorofor är inmärkt i probens 3’-ände och kallas donatorfluorofor. Den andre probens fluorofor är inmärkt i dess 5’-ände och kallas mottagarfluorofor. Principen för detektion är att proberna binder in till mål-DNA och donatorfluoroforen exiteras av realtids-PCR-instrumentet, dess emissionsenergi överförs till mottagarfluoroforen som blir exiterad och en tredje energi frigörs från mottagarfluoroforen som tas upp av detektorn i realtids-PCR-instrumentet ('''figur 10'''). Denna energiövergång fungerar inte i fri lösning då proberna inte är hybridiserade nära varandra. I PCR-reaktionens denatureringsfas smälter, lossnar, proberna från mål-DNA och kan återanvändas, till skillnad från en TaqMan-probe som hydrolyseras. Efter PCR-reaktionen kan man utföra en smältanalys för att få fram smältvärdet för proberna till amplikonet. Man kan utnyttja smältanalysen för att särskilja olika genotyper som exempel för [[adenovirus]]. I detta exempel amplifieras och detekteras både serotyp 40 och 41 av samma primerpar och prober men vid smältanalys får de olika värden beroende på serotyp. Detta beror på att proberna binder olika hårt till de två serotypamplikonen då en av serotyperna har en eller flera mis-match där proben hybridiserar dvs binder. | Första exemplet på en hybridiseringsprobe är ''Fluorescence Resonance Energy Transfer''-probe som förkortas ''FRET''-probe. Den består av två oligonukleotider inmärkta med varsin fluorofor men med olika excitations- och emissionsspektra. Proberna är designade så att de hybridiserar, binder in, till mål-DNA med endast några basers mellanrum. Den första probens fluorofor är inmärkt i probens 3’-ände och kallas donatorfluorofor. Den andre probens fluorofor är inmärkt i dess 5’-ände och kallas mottagarfluorofor. Principen för detektion är att proberna binder in till mål-DNA och donatorfluoroforen exiteras av realtids-PCR-instrumentet, dess emissionsenergi överförs till mottagarfluoroforen som blir exiterad och en tredje energi frigörs från mottagarfluoroforen som tas upp av detektorn i realtids-PCR-instrumentet ('''figur 10'''). Denna energiövergång fungerar inte i fri lösning då proberna inte är hybridiserade nära varandra. I PCR-reaktionens denatureringsfas smälter, lossnar, proberna från mål-DNA och kan återanvändas, till skillnad från en TaqMan-probe som hydrolyseras. Efter PCR-reaktionen kan man utföra en smältanalys för att få fram smältvärdet för proberna till amplikonet. Man kan utnyttja smältanalysen för att särskilja olika genotyper som exempel för [[adenovirus]]. I detta exempel amplifieras och detekteras både serotyp 40 och 41 av samma primerpar och prober men vid smältanalys får de olika värden beroende på serotyp. Detta beror på att proberna binder olika hårt till de två serotypamplikonen då en av serotyperna har en eller flera mis-match där proben hybridiserar dvs binder. | ||
| − | [[Fil:molekylfig10.jpg | + | [[Fil:molekylfig10.jpg|thumb|center|500px||'''Figur 10'''. FRET-probe. (A) Donatorproben i fri form avger energi medan mottagarproben (acceptor) inte avger någon energi. (B) Under annealingsteget binder båda prober in och energin från donatorproben överförs till mottagarproben som exiteras och vars emissionsenergi kan detekteras i PCR-instrumentet. (C) När proberna avlägsnas sker ingen energiöverföring mellan proberna och signalen släcks ned. ''Bild: © QIAGEN, all rights reserved'']] |
| − | |||
| − | '''Figur 10'''. FRET-probe. (A) Donatorproben i fri form avger energi medan mottagarproben (acceptor) inte avger någon energi. (B) Under annealingsteget binder båda prober in och energin från donatorproben överförs till mottagarproben som exiteras och vars emissionsenergi kan detekteras i PCR-instrumentet. (C) När proberna avlägsnas sker ingen energiöverföring mellan proberna och signalen släcks ned. ''Bild: © QIAGEN, all rights reserved'' | ||
=== Molecular beacon-probe === | === Molecular beacon-probe === | ||
| Rad 37: | Rad 33: | ||
''Molecular beacon-probe'' (beacon betyder fyr) är ett annat exempel på hybridiseringsprobe. Proben har två specifika strukturella former, en stam och en ögla ('''figur 11'''). Stamstrukturen hålles samman genom hybridisering då kvävebaserna är så valda att de basparar till varandra i fri lösning. Vardera 5’- respektive 3’-ändarna är inmärkta med en reporterfluorofor respektive quencher. Specificiteten för molecular beacon-proben ligger i öglans nukleinsyrasekvens vilken är komplementär till mål-DNA. Under PCR-reaktionen smälter stamstrukturen isär och om öglestrukturen hybridiserar till mål-DNA separeras reporter och quencher fysiskt. Därvid exiteras reportern dess emissionsenergi tas upp av detektorn i realtids-PCR-maskinen. På samma sätt som FRET-probe kan den återanvändas efter denaturering. | ''Molecular beacon-probe'' (beacon betyder fyr) är ett annat exempel på hybridiseringsprobe. Proben har två specifika strukturella former, en stam och en ögla ('''figur 11'''). Stamstrukturen hålles samman genom hybridisering då kvävebaserna är så valda att de basparar till varandra i fri lösning. Vardera 5’- respektive 3’-ändarna är inmärkta med en reporterfluorofor respektive quencher. Specificiteten för molecular beacon-proben ligger i öglans nukleinsyrasekvens vilken är komplementär till mål-DNA. Under PCR-reaktionen smälter stamstrukturen isär och om öglestrukturen hybridiserar till mål-DNA separeras reporter och quencher fysiskt. Därvid exiteras reportern dess emissionsenergi tas upp av detektorn i realtids-PCR-maskinen. På samma sätt som FRET-probe kan den återanvändas efter denaturering. | ||
| − | [[Fil:molekylfig11.jpg | + | [[Fil:molekylfig11.jpg|thumb|center|500px||'''Figur 11'''. Molecular beacon-probe. (A) Molecular beacon-probe i fri form är reporter (fluorofor) och quencher nära varandra och all energi från fluoroforen tas upp av quenchern. (B) När proben bundit till templatet skiljs fluorofor och quencher åt så fluoroforens emissionsenergi kan detekteras av PCR-instrumentet. (C) När proben avlägsnas återgår den till sitt ursprung i form av en hårnåls struktur. ''Bild: © QIAGEN, all rights reserved'']] |
| − | |||
| − | '''Figur 11'''. Molecular beacon-probe. (A) Molecular beacon-probe i fri form är reporter (fluorofor) och quencher nära varandra och all energi från fluoroforen tas upp av quenchern. (B) När proben bundit till templatet skiljs fluorofor och quencher åt så fluoroforens emissionsenergi kan detekteras av PCR-instrumentet. (C) När proben avlägsnas återgår den till sitt ursprung i form av en hårnåls struktur. ''Bild: © QIAGEN, all rights reserved'' | ||
=== SYBR Green === | === SYBR Green === | ||
| Rad 48: | Rad 42: | ||
| − | [[Fil:molekylfig12.jpg]] | + | [[Fil:molekylfig12.jpg|thumb|center|500px|||'''Figur 12'''. SYBR Green. Principen för SYBR Green I baserad detektion är att ju mer SYBR Green I som binder in till dubbelsträngat DNA ju mer emissionsenergi avges. ''Bild: © QIAGEN, all rights reserved'']] |
| − | |||
| − | |||
Efter avslutad amplifiering utförs i realtids-PCR-instrumentet en smältpunktsanalys som kan skilja mellan amplikonet och eventuella biprodukter ('''figur 13'''). Smältpunkten hos dubbelsträngat DNA beror på sekvensens längd och nukleinsyrasammansättningen vilket gör varje amplikon specifikt gällande smältpunkt. Smältpunktsanalys utförs genom att temperaturen gradvis ökas med några tiondels grader per sekund och fluorescensen mäts kontinuerligt. Smältpunkten definieras som den temperatur där hälften av DNA-strängarna har separerats och därmed hälften av SYBR Green I molekylerna lossnat från DNA. Detta kan förtydligas med beräkning av den negativa derivatan av funktionen fluorescensintensitet mot temperaturen där smältpunkten motsvarar en topp i diagrammet. Ett sätt att öka specificiteten hos SYBR Green I under en PCR-reaktion är att utföra mätning av emissionsenergin vid en temperatur (Tmätning) som är under smältpunkten för amplikonet (TmPCR-produkt) men högre än smältpunkten för tex primer-dimer (Tmprimer-dimer) enligt följande Tmprimer-dimer <Tmätning < TmPCR-produkt. Det man uppnår är att man mäter amplifiering av endast amplikonet och inte summan av biprodukt plus amplikon. En tumregel är att Tmätning ligger ungefär 5°C under TmPCR-produkt. Smältpunktsanalys kan också genomföras med hydrolysprober som FRET-probe och molecular beacon-probe där varje probes smältpunkt är specifik och man kan skilja på olika prober i samma PCR-reaktion, vid tex multiplex PCR. | Efter avslutad amplifiering utförs i realtids-PCR-instrumentet en smältpunktsanalys som kan skilja mellan amplikonet och eventuella biprodukter ('''figur 13'''). Smältpunkten hos dubbelsträngat DNA beror på sekvensens längd och nukleinsyrasammansättningen vilket gör varje amplikon specifikt gällande smältpunkt. Smältpunktsanalys utförs genom att temperaturen gradvis ökas med några tiondels grader per sekund och fluorescensen mäts kontinuerligt. Smältpunkten definieras som den temperatur där hälften av DNA-strängarna har separerats och därmed hälften av SYBR Green I molekylerna lossnat från DNA. Detta kan förtydligas med beräkning av den negativa derivatan av funktionen fluorescensintensitet mot temperaturen där smältpunkten motsvarar en topp i diagrammet. Ett sätt att öka specificiteten hos SYBR Green I under en PCR-reaktion är att utföra mätning av emissionsenergin vid en temperatur (Tmätning) som är under smältpunkten för amplikonet (TmPCR-produkt) men högre än smältpunkten för tex primer-dimer (Tmprimer-dimer) enligt följande Tmprimer-dimer <Tmätning < TmPCR-produkt. Det man uppnår är att man mäter amplifiering av endast amplikonet och inte summan av biprodukt plus amplikon. En tumregel är att Tmätning ligger ungefär 5°C under TmPCR-produkt. Smältpunktsanalys kan också genomföras med hydrolysprober som FRET-probe och molecular beacon-probe där varje probes smältpunkt är specifik och man kan skilja på olika prober i samma PCR-reaktion, vid tex multiplex PCR. | ||
| Rad 57: | Rad 49: | ||
High-resolution melting, HRM, är en utveckling av smältpunktsanalys där man kan få fram mer detaljerad information om amplikonets sammansättning. Metoden används för att upptäcka genetiska variationer som tex SNP (singel nucleotide polymorphism) och mutationer vilket medför att ett användningsområde är typning av bakterier och virus. Man kan även använda tekniken till att upptäcka nya, okända varianter, sk gene scanning. Metoden upptäcker en blandning av olika amplikon med en liten skillnad i bas-sammansättningen (tex SNP) vilket visas som högupplösta smältkurvor. Mängdförhållandet mellan olika amplikon kan också skilja mycket men framkommer i analysen. Metoden kräver PCR-maskiner med analysprogram utvecklade för HRM. SYBR Green fungerar till viss del som analysfärg men det finns specifikt utvecklade färger för HRM-analys, exempelvis Eva Green, LC Green och ResoLight. HRM är kostnadsmässigt en billigare metod än probetekniken. | High-resolution melting, HRM, är en utveckling av smältpunktsanalys där man kan få fram mer detaljerad information om amplikonets sammansättning. Metoden används för att upptäcka genetiska variationer som tex SNP (singel nucleotide polymorphism) och mutationer vilket medför att ett användningsområde är typning av bakterier och virus. Man kan även använda tekniken till att upptäcka nya, okända varianter, sk gene scanning. Metoden upptäcker en blandning av olika amplikon med en liten skillnad i bas-sammansättningen (tex SNP) vilket visas som högupplösta smältkurvor. Mängdförhållandet mellan olika amplikon kan också skilja mycket men framkommer i analysen. Metoden kräver PCR-maskiner med analysprogram utvecklade för HRM. SYBR Green fungerar till viss del som analysfärg men det finns specifikt utvecklade färger för HRM-analys, exempelvis Eva Green, LC Green och ResoLight. HRM är kostnadsmässigt en billigare metod än probetekniken. | ||
| − | [[Fil:molekylfig13.jpg | + | [[Fil:molekylfig13.jpg|thumb|center|600px||'''Figur 13.''' Smältanalys. (A) Vid låg temperatur är all SYBR Green I inbundet till intakt dubbelsträngat DNA och mycket fluorescens kan detekteras. (B) Smältpunkten (Tm) för PCR-produkten är när hälften av DNA-strängarna separerats och hälften av SYBR Green I molekylerna är i fri form. Detta illustreras i den nedre kurvan (negativa derivatan av den övre kurvan) som en topp, smälttopp. (C) Alla DNA-strängar är separerade och ingen energi avges från SYBR Green I. ''Bild: Fredrik Aronsson'']] |
| − | |||
| − | '''Figur 13.''' Smältanalys. (A) Vid låg temperatur är all SYBR Green I inbundet till intakt dubbelsträngat DNA och mycket fluorescens kan detekteras. (B) Smältpunkten (Tm) för PCR-produkten är när hälften av DNA-strängarna separerats och hälften av SYBR Green I molekylerna är i fri form. Detta illustreras i den nedre kurvan (negativa derivatan av den övre kurvan) som en topp, smälttopp. (C) Alla DNA-strängar är separerade och ingen energi avges från SYBR Green I. ''Bild: Fredrik Aronsson'' | ||
=== LUX primer === | === LUX primer === | ||
| Rad 66: | Rad 56: | ||
| − | [[Fil:molekylfig14.jpg | + | [[Fil:molekylfig14.jpg|thumb|center|600px||||'''Figur 14'''. LUX primer. Principen för LUX primer är att den i sin ursprungsform, hårnål, avger lite energi medan inbundet som dubbelsträngat DNA avger mycket energi. Ju fler amplikon som LUX primern inkorporerats i desto mer energi kan detekteras i PCR-instrumentet. ''Bild: Invitrogen, publicerad med tillstånd från företaget''.]] |
| − | |||
| − | '''Figur 14'''. LUX primer. Principen för LUX primer är att den i sin ursprungsform, hårnål, avger lite energi medan inbundet som dubbelsträngat DNA avger mycket energi. Ju fler amplikon som LUX primern inkorporerats i desto mer energi kan detekteras i PCR-instrumentet. ''Bild: Invitrogen, publicerad med tillstånd från företaget''. | ||
== Kvalitativ och kvantitativ realtids-PCR analys == | == Kvalitativ och kvantitativ realtids-PCR analys == | ||
| Rad 78: | Rad 66: | ||
''Kvantitativ realtids-PCR'' bygger på att man har en hög effektivitet i PCR-reaktionen vilket kan beräknas enligt formeln 1/log (effektivitet) = cykel/log [DNA]. Vid effektiviteten 2.0 dvs 100 % så ökar cykeltalet med 3.32 cykler vid en spädningsserie på 1:10. Effektiviteten minskar oftast vid höga Ct-värden när färre starttemplat finns tillgängliga för PCR-reaktionen. Därför begränsar man ofta den kvantitativa realtids-PCR till ett område där effektiviteten är hög och med det en större säkerhet i svaret. Värden utanför området tillskriver man mer än eller mindre än metodens mätbara antal kopior/mL. | ''Kvantitativ realtids-PCR'' bygger på att man har en hög effektivitet i PCR-reaktionen vilket kan beräknas enligt formeln 1/log (effektivitet) = cykel/log [DNA]. Vid effektiviteten 2.0 dvs 100 % så ökar cykeltalet med 3.32 cykler vid en spädningsserie på 1:10. Effektiviteten minskar oftast vid höga Ct-värden när färre starttemplat finns tillgängliga för PCR-reaktionen. Därför begränsar man ofta den kvantitativa realtids-PCR till ett område där effektiviteten är hög och med det en större säkerhet i svaret. Värden utanför området tillskriver man mer än eller mindre än metodens mätbara antal kopior/mL. | ||
| − | [[Fil:molekylfig15.jpg]] | + | [[Fil:molekylfig15.jpg|thumb|center|600px||'''Figur 15'''. Amplifierningskurva. När amplifieringskurvan passerar tröskeln kan man avläsa cykeltröskelvärdet (Ct-värdet) för en positiv PCR-reaktion. När man jämför Ct-värden innebär ett lägre Ct-värde att det finns mer starttemplat, DNA/RNA, i provet jämfört med ett Ct-värde som är högre. ''Bild: Fredrik Aronsson'']] |
| − | + | ||
[[Kategori:Molekylärbiologisk diagnostik]] | [[Kategori:Molekylärbiologisk diagnostik]] | ||
Nuvarande version från 1 september 2010 kl. 15.31
Till innehållsförteckningen för Referensmetodik:Molekylärbiologisk diagnostik
Realtids-PCR[redigera]
Den konventionella PCR-tekniken har vidareutvecklats så att detektion av PCR-produkten sker direkt i PCR-röret och amplifieringen kan följas i realtid. Det finns stora diagnostiska vinster med realtids-PCR jämfört med konventionell PCR. En stor fördel är att kontamineringsrisken, carry over, minskar drastiskt då amplifierat material inte behöver tas upp ur röret. Efter analys kasseras PCR-produkten i ett slutet rör eller platta. Tidsåtgång och arbetsmoment minskar genom att detektionssteget är inbyggt i PCR-reaktionen och analyssteg som elektrofores eller blottning inte är nödvändiga. Vid realtids-PCR sker detektionen av PCR-produkten med hjälp av fluorescerande molekyler vilket ökar känsligheten i metoden jämfört med traditionell detektion med exempelvis etidiumbromid. De fluorescerande molekyler som används kan antingen vara i fri form, vanligtvis SYBR green I, eller bundna till en probe, t ex TaqMan-probe. Fluorescenssignalen som bildas är proportionell mot mängden amplifierat DNA i realtids-PCR-reaktionen vilket medför att man kan kvantifiera nukleinsyrainnehållet i sitt ursprungsprov.
Nedre detektionsnivå är teoretiskt en kopia av templat-DNA men det finns ingen garanti för att denna enda kopia kommer att delta i PCR-reaktionen. Om man i ett patientprov har en koncentration av templatet som innebär att det till PCR-reaktionen i genomsnitt kommer att överföras 2 kopior/reaktion kommer det i ungefär 10 % av PCR-reaktionerna att saknas ett starttemplat. Om medeltalet istället är 5 kopior/reaktion kommer 99 % av PCR-reaktionerna att ha minst 1 startkopia och med i medeltal 10 kopior/reaktion minskas risken till 0,01 % för falskt negativa resultat.
Probeteknik[redigera]
Som beskrivits tidigare under PCR allmänt och figur 8 omges den vid PCR amplifierade sekvensen av två primrar. En probe (hybridiseringssond, ibland kallat sökfragment) för realtids-PCR är lokaliserad uppströms den ena primern och inom amplifieringsområdet. Proben består av en oligonukleotid, precis som primern, men är inmärkt med en fluorofor och/eller quencher, ”släckare”. Man skiljer vanligtvis mellan en hydrolysprobe (tex TaqMan-probe) och hybridiseringsprobe (tex FRET-probe eller Molecular beacon-probe). Genom att använda probeteknik vid realtids-PCR ökar man specificiteten för PCR-reaktionen. Samtidigt ökar risken för falskt negativa resultat i de fall mutationer skulle ha uppstått i bindningsområdet för proben i mål-DNA-molekylen.
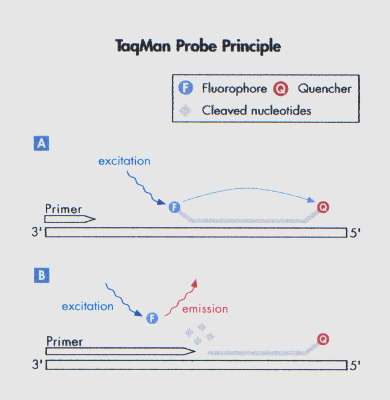
TaqMan-probe[redigera]
Den vanligaste typen av hydrolysprobe är TaqMan-probe. Den är i sin 5’-ände inmärkt med en fluorofor, en så kallad reporter och i sin 3’-ände inmärkt med en quencher. Vid amplifiering binder proben in till mål-DNA och under elongeringsfasen hydrolyseras proben, bryts ned, av Taq-polymerasets 5’-exonukleas aktivitet varvid reporter och quencher skiljs åt (figur 9). Energin från den fria och exciterade reportern absorberas inte längre av quenchern utan tas nu upp av detektorn i realtids-PCR-instrumentet. Specificiteten för en TaqMan-probe kan ökas genom att man i 3’-änden fäster en så kallad minorgroove binder (MGB)-molekyl. PCR-reaktionen blir nu mer robust då MGB-molekylen binder hårdare till DNA-molekylen, vilket innebär att probens smältvärde (Tm) ökar. Hydrolysprober fungerar bäst vid tvåstegs-PCR, vilket innebär att annealing och elongeringsfaserna slås samman till en fas i PCR-reaktionen.
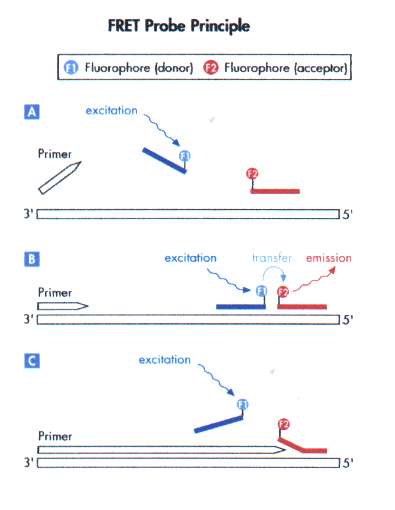
FRET-probe[redigera]
Första exemplet på en hybridiseringsprobe är Fluorescence Resonance Energy Transfer-probe som förkortas FRET-probe. Den består av två oligonukleotider inmärkta med varsin fluorofor men med olika excitations- och emissionsspektra. Proberna är designade så att de hybridiserar, binder in, till mål-DNA med endast några basers mellanrum. Den första probens fluorofor är inmärkt i probens 3’-ände och kallas donatorfluorofor. Den andre probens fluorofor är inmärkt i dess 5’-ände och kallas mottagarfluorofor. Principen för detektion är att proberna binder in till mål-DNA och donatorfluoroforen exiteras av realtids-PCR-instrumentet, dess emissionsenergi överförs till mottagarfluoroforen som blir exiterad och en tredje energi frigörs från mottagarfluoroforen som tas upp av detektorn i realtids-PCR-instrumentet (figur 10). Denna energiövergång fungerar inte i fri lösning då proberna inte är hybridiserade nära varandra. I PCR-reaktionens denatureringsfas smälter, lossnar, proberna från mål-DNA och kan återanvändas, till skillnad från en TaqMan-probe som hydrolyseras. Efter PCR-reaktionen kan man utföra en smältanalys för att få fram smältvärdet för proberna till amplikonet. Man kan utnyttja smältanalysen för att särskilja olika genotyper som exempel för adenovirus. I detta exempel amplifieras och detekteras både serotyp 40 och 41 av samma primerpar och prober men vid smältanalys får de olika värden beroende på serotyp. Detta beror på att proberna binder olika hårt till de två serotypamplikonen då en av serotyperna har en eller flera mis-match där proben hybridiserar dvs binder.
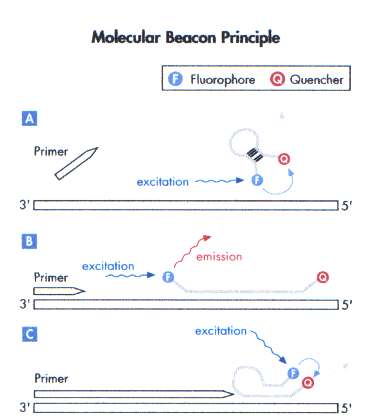
Molecular beacon-probe[redigera]
Molecular beacon-probe (beacon betyder fyr) är ett annat exempel på hybridiseringsprobe. Proben har två specifika strukturella former, en stam och en ögla (figur 11). Stamstrukturen hålles samman genom hybridisering då kvävebaserna är så valda att de basparar till varandra i fri lösning. Vardera 5’- respektive 3’-ändarna är inmärkta med en reporterfluorofor respektive quencher. Specificiteten för molecular beacon-proben ligger i öglans nukleinsyrasekvens vilken är komplementär till mål-DNA. Under PCR-reaktionen smälter stamstrukturen isär och om öglestrukturen hybridiserar till mål-DNA separeras reporter och quencher fysiskt. Därvid exiteras reportern dess emissionsenergi tas upp av detektorn i realtids-PCR-maskinen. På samma sätt som FRET-probe kan den återanvändas efter denaturering.
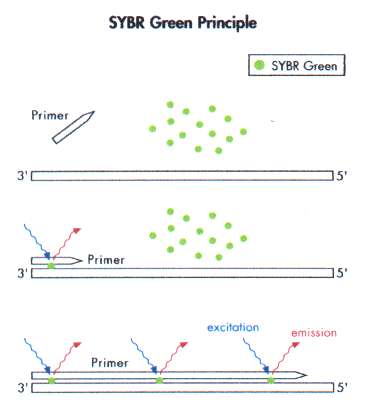
SYBR Green[redigera]
SYBR Green I är en cyaninfärg som är specifik för dubbelsträngat DNA och binder in till minorgroove i DNA-molekylens α-helix. Endast inbundet SYBR Green I kan exiteras och avge emissionsenergi (figur 12). Samtidigt är inbindningen ospecifik då SYBR Green I binder in till allt dubbelsträngat DNA. Om en PCR-reaktion inte är fullständigt optimerad binder SYBR Green I till, förutom önskat amplikon, även till biprodukter som tex primer-dimer.
Efter avslutad amplifiering utförs i realtids-PCR-instrumentet en smältpunktsanalys som kan skilja mellan amplikonet och eventuella biprodukter (figur 13). Smältpunkten hos dubbelsträngat DNA beror på sekvensens längd och nukleinsyrasammansättningen vilket gör varje amplikon specifikt gällande smältpunkt. Smältpunktsanalys utförs genom att temperaturen gradvis ökas med några tiondels grader per sekund och fluorescensen mäts kontinuerligt. Smältpunkten definieras som den temperatur där hälften av DNA-strängarna har separerats och därmed hälften av SYBR Green I molekylerna lossnat från DNA. Detta kan förtydligas med beräkning av den negativa derivatan av funktionen fluorescensintensitet mot temperaturen där smältpunkten motsvarar en topp i diagrammet. Ett sätt att öka specificiteten hos SYBR Green I under en PCR-reaktion är att utföra mätning av emissionsenergin vid en temperatur (Tmätning) som är under smältpunkten för amplikonet (TmPCR-produkt) men högre än smältpunkten för tex primer-dimer (Tmprimer-dimer) enligt följande Tmprimer-dimer <Tmätning < TmPCR-produkt. Det man uppnår är att man mäter amplifiering av endast amplikonet och inte summan av biprodukt plus amplikon. En tumregel är att Tmätning ligger ungefär 5°C under TmPCR-produkt. Smältpunktsanalys kan också genomföras med hydrolysprober som FRET-probe och molecular beacon-probe där varje probes smältpunkt är specifik och man kan skilja på olika prober i samma PCR-reaktion, vid tex multiplex PCR.
High-resolution melting, HRM, är en utveckling av smältpunktsanalys där man kan få fram mer detaljerad information om amplikonets sammansättning. Metoden används för att upptäcka genetiska variationer som tex SNP (singel nucleotide polymorphism) och mutationer vilket medför att ett användningsområde är typning av bakterier och virus. Man kan även använda tekniken till att upptäcka nya, okända varianter, sk gene scanning. Metoden upptäcker en blandning av olika amplikon med en liten skillnad i bas-sammansättningen (tex SNP) vilket visas som högupplösta smältkurvor. Mängdförhållandet mellan olika amplikon kan också skilja mycket men framkommer i analysen. Metoden kräver PCR-maskiner med analysprogram utvecklade för HRM. SYBR Green fungerar till viss del som analysfärg men det finns specifikt utvecklade färger för HRM-analys, exempelvis Eva Green, LC Green och ResoLight. HRM är kostnadsmässigt en billigare metod än probetekniken.
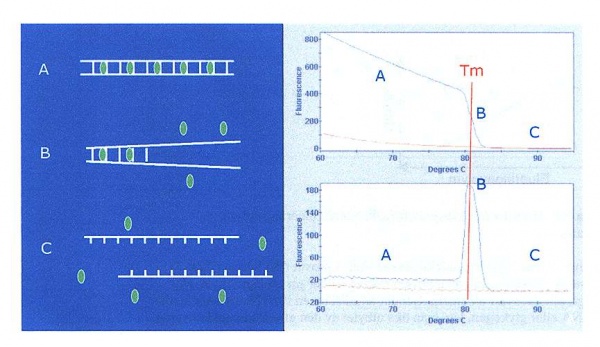
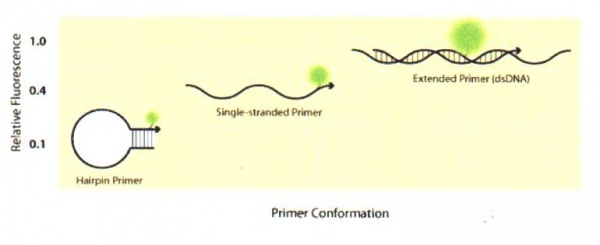
LUX primer[redigera]
LUX står för light upon extension . Den ena primern i primerparet har i fri lösning formen av en hårnål (figur 14) vilket innebär att primern får en stamstruktur då den hybridiserar till sig själv. Primern är inmärkt med en fluorofor nära 3’-änden i stamstrukturen och är 20-30 baser i längd. När den är i denna form är fluoroforen släckt och det behövs ingen separat quencher för att släcka den. När primern är inkorporerad i dubbelsträngat DNA i PCR-produkten ökar fluorescenssignalen från LUX primern vilket kan detekteras i realtids-PCR-instrumentet. Fördelen med denna teknik är att det inte behövs någon probe för att uppnå hög känslighet vilket också medför en lägre kostnad. Specificiteten minskar något jämfört med en probe men att designa ett primerpar är enklare än att designa både ett primerpar och en probe. Med LUX-tekniken har man även möjlighet att genomföra en smältpunktsanalys för amplikonverifiering.
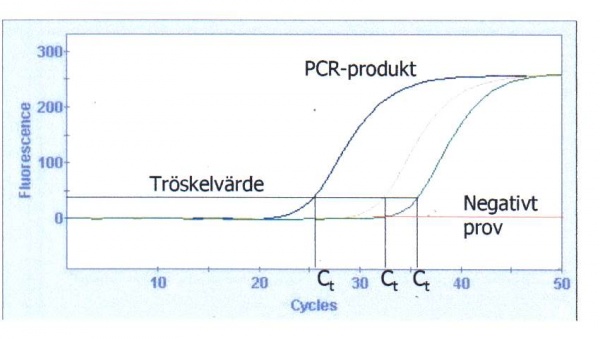
Kvalitativ och kvantitativ realtids-PCR analys[redigera]
Vid en realtids-PCR-analys erhålls en amplifieringskurva för positiva PCR-reaktioner (figur 15). Antalet amplikon ökar exponentiellt och därmed signalen tills en platåfas nås vilket främst beror på att aktiviteten hos Taq-polymeraset är förbrukad men även på att fria nukleotider, primers och probe tar slut eller på att PCR-produkter finns i sådant överskott att DNA-strängarna i dessa hittar och hybridiserar till varandra oftare än till primer och probe. I analysen lägger man in eller har förprogrammerat ett tröskelvärde vilket motsvarar den fluorescensnivå som tillväxtkurvan ska passera för att tolka provet som positivt. Vid den punkt där amplifieringskurvan skär tröskelvärdet kan man på x-axeln avläsa cykeltröskelvärdet (Ct-värde) för det positiva provet. Vid kvalitativ analys vill man påvisa eller inte påvisa sökt nukleinsyra. Påvisas nukleinsyran får man ett Ct-värde, annars inte.
Ett prov med lågt Ct-värde innehåller från början fler starttemplat jämfört med ett prov med högre Ct-värde. Detta utnyttjas vid kvantitativ analys där prover med okänd koncentration jämförs mot en standardkurva baserad på ett antal standardprov som analyseras samtidigt med proverna. Det är viktigt att tröskeln passeras i tillväxtfasen, logfasen, av amplifieringskurvan för att få ett korrekt Ct-värde. Om man statistiskt kan visa att standardkurvan inte förändras mellan olika PCR-analyser behöver man inte analysera standardprover vid varje analystillfälle. Vid detta tillfälle använder man då en färdig standardkurva i analysmjukvaran. När man jämför Ct-värdet mellan olika PCR-analyser och saknar en standardkurva är det viktigt att tröskeln har ett fast värde mellan de olika analyserna.
Kvantitativ realtids-PCR bygger på att man har en hög effektivitet i PCR-reaktionen vilket kan beräknas enligt formeln 1/log (effektivitet) = cykel/log [DNA]. Vid effektiviteten 2.0 dvs 100 % så ökar cykeltalet med 3.32 cykler vid en spädningsserie på 1:10. Effektiviteten minskar oftast vid höga Ct-värden när färre starttemplat finns tillgängliga för PCR-reaktionen. Därför begränsar man ofta den kvantitativa realtids-PCR till ett område där effektiviteten är hög och med det en större säkerhet i svaret. Värden utanför området tillskriver man mer än eller mindre än metodens mätbara antal kopior/mL.
REFERENSER[redigera]
- 1. Molecular microbiology: diagnostic principles and practice / editors, David H Persing et al. ASM press, Washington DC, 2004.
- 2. The polymerase chain reaction / editors Kary B Mullis et al. Birkhäuser, Boston, 1994.
- 3. Nazarenko, I et al. Nucleic Acids Research 2002; 30(9): e37.
- 4. Jothikumar, N. et al. Quantitative Real-Time PCR Assays for Detection of Human Adenoviruses and Identification of Serotype 40 and 41. Appl Environ Microbiol 2005; 71:3131-3136.