Skillnad mellan versioner av "Sekvensering"
(→SOLiD) |
|||
| (17 mellanliggande sidversioner av samma användare visas inte) | |||
| Rad 6: | Rad 6: | ||
DNA-sekvensering är den process som används för att med biokemiska metoder bestämma ordningen av baserna adenin, guanin, cytosin och tymin i [[Arvsmassans struktur och funktion|DNA]]. Sekvensen utgör den ärvda genetiska informationen och är därför viktig både i grundläggande forskning kring organismer, diagnostik, vid rättsmedicinska utredningar för att identifiera ärftliga sjukdomar och vid epidemiologiska utredningar. Den snabba utvecklingen inom området har möjliggjort storskaliga sekvenseringar av till exempel det mänskliga genomet. Med de nya sekvenseringsteknikerna får man längre och fler sekvenser med färre fel på kortare tid. Utvecklingen av enzymer, instrument och programvara pågår ständigt och de uppgifter på kapacitet man uppger idag är föråldrade inom kort. | DNA-sekvensering är den process som används för att med biokemiska metoder bestämma ordningen av baserna adenin, guanin, cytosin och tymin i [[Arvsmassans struktur och funktion|DNA]]. Sekvensen utgör den ärvda genetiska informationen och är därför viktig både i grundläggande forskning kring organismer, diagnostik, vid rättsmedicinska utredningar för att identifiera ärftliga sjukdomar och vid epidemiologiska utredningar. Den snabba utvecklingen inom området har möjliggjort storskaliga sekvenseringar av till exempel det mänskliga genomet. Med de nya sekvenseringsteknikerna får man längre och fler sekvenser med färre fel på kortare tid. Utvecklingen av enzymer, instrument och programvara pågår ständigt och de uppgifter på kapacitet man uppger idag är föråldrade inom kort. | ||
| − | |||
=== Historik === | === Historik === | ||
| Rad 18: | Rad 17: | ||
Under tidsperiod 1975-1977 utvecklade ''Sanger'' en sekvenseringsmetod för enkelsträngat DNA. Huvudprincipen bakom Sangers metod är användandet av dideoxy-nukleotider som orsakar stopp i DNA-syntesen, se '''figur 18'''. På 70- och 80-talen krävdes det att man framställde enkelsträngat DNA innan sekvensering med dideoxy-metoden kunde utföras. Oftast klonade man in det fragment man skulle sekvensera i en bakteriofag (M13) som har både en enkelsträngad och dubbelsträngad fas i sin replikationscykel. Man använde den dubbelsträngade DNA-formen för att klona in DNA-fragmentet. Sedan anrikade man den enkelsträngade formen för sekvenseringen. När PCR-metoden introducerades i slutet av 80-talet blev det möjligt att på ett enkelt sätt sekvensera med dideoxy-metoden. Med hjälp av PCR kan man amplifiera och analysera små mängder av templat. Med hjälp av färginmärkning av dideoxynukleotiderna separeras sekvenseringsprodukterna i kapillärer istället för stora gelelektroforessystem som tidigare användes ('''figur 19'''). Med all metodutveckling kring templatframställning, enzymer, färginmärkning och gel/kapillärseparering kan man idag läsa mellan 500-1000 långa fragment för upp till 384 prover samtidigt. Den ökande mängden sekvensdata har även krävt utveckling av dataprogrammen/mjukvaran för hantering av all sekvensinformation. | Under tidsperiod 1975-1977 utvecklade ''Sanger'' en sekvenseringsmetod för enkelsträngat DNA. Huvudprincipen bakom Sangers metod är användandet av dideoxy-nukleotider som orsakar stopp i DNA-syntesen, se '''figur 18'''. På 70- och 80-talen krävdes det att man framställde enkelsträngat DNA innan sekvensering med dideoxy-metoden kunde utföras. Oftast klonade man in det fragment man skulle sekvensera i en bakteriofag (M13) som har både en enkelsträngad och dubbelsträngad fas i sin replikationscykel. Man använde den dubbelsträngade DNA-formen för att klona in DNA-fragmentet. Sedan anrikade man den enkelsträngade formen för sekvenseringen. När PCR-metoden introducerades i slutet av 80-talet blev det möjligt att på ett enkelt sätt sekvensera med dideoxy-metoden. Med hjälp av PCR kan man amplifiera och analysera små mängder av templat. Med hjälp av färginmärkning av dideoxynukleotiderna separeras sekvenseringsprodukterna i kapillärer istället för stora gelelektroforessystem som tidigare användes ('''figur 19'''). Med all metodutveckling kring templatframställning, enzymer, färginmärkning och gel/kapillärseparering kan man idag läsa mellan 500-1000 långa fragment för upp till 384 prover samtidigt. Den ökande mängden sekvensdata har även krävt utveckling av dataprogrammen/mjukvaran för hantering av all sekvensinformation. | ||
| − | |||
| − | |||
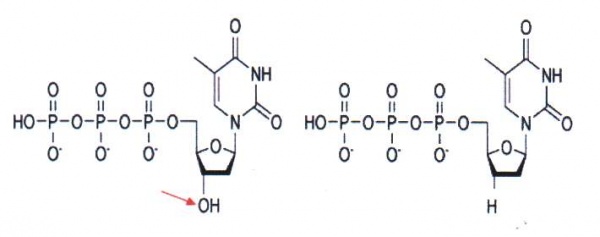
| + | [[Fil:molekylfig18b.jpg|thumb|center|600px| | '''Figur 18'''. Skillnaden mellan deoxytymidintrifosfat (dTTP) och dideoxytymidintrifosfat (ddTTP, till höger i bilden) är avsaknaden av syret på kol 3 i ribos. Ingen fortsatt DNA syntes kan ske från dideoxynukleotider. ''Bild: Mia Brytting'']] | ||
| − | + | [[Fil:molekylfig19b.jpg|thumb|center|600px| '''Figur 19'''. Sekvensering, ''Sangers'' metod. DNA (templat) som ska sekvenseras blandas med en mindre mängd fluorofor (färg)- inmärkta dideoxy-nukleotider (ddNTP: en fluorofor för varje ddATP, ddCTG, ddGTG samt ddTTP), vanliga trifosfatnukleotider, ett DNA-polymeras, en primer och därefter startas PCR-programmet i en termocykler. Dideoxynukleotiderna är derivat av normala trifosfatnukleotider, och saknar både OH-grupp på 2'-kolet men även på 3'-kolet. Eftersom dideoxynukleotiderna är färre till antalet än de vanliga nukleotiderna är det högre sannolikhet att en vanlig nukleotid inkorporeras, men när en dideoxy-nukleotid byggs in av DNA-polymeraset stoppas DNA-syntesen eftersom OH-grupp saknas på 3'-kolet. Efter sekvenseringen har man en produkt som består av olika längder. Genom att separera DNA-fragmenten beroende av deras längd i en maskin som kan läsa av de olika fluoroforernas färger kan DNA sekvensen läsas direkt. ''Bild: Mia Brytting'']] | |
| − | '''Figur 19'''. Sekvensering, ''Sangers'' metod. DNA (templat) som ska sekvenseras blandas med en mindre mängd fluorofor (färg)- inmärkta dideoxy-nukleotider (ddNTP: en fluorofor för varje ddATP, ddCTG, ddGTG samt ddTTP), vanliga trifosfatnukleotider, ett DNA-polymeras, en primer och därefter startas PCR-programmet i en termocykler. Dideoxynukleotiderna är derivat av normala trifosfatnukleotider, och saknar både OH-grupp på 2'-kolet men även på 3'-kolet. Eftersom dideoxynukleotiderna är färre till antalet än de vanliga nukleotiderna är det högre sannolikhet att en vanlig nukleotid inkorporeras, men när en dideoxy-nukleotid byggs in av DNA-polymeraset stoppas DNA-syntesen eftersom OH-grupp saknas på 3'-kolet. Efter sekvenseringen har man en produkt som består av olika längder. Genom att separera DNA-fragmenten beroende av deras längd i en maskin som kan läsa av de olika fluoroforernas färger kan DNA sekvensen läsas direkt. ''Bild: Mia Brytting'' | ||
| − | |||
=== Pyrosekvensering === | === Pyrosekvensering === | ||
| Rad 32: | Rad 28: | ||
| − | + | [[Fil:molekylfig20.jpg|thumb|center|500px| '''Figur 20.''' ''Pyrosekvensering''. Metoden bygger på att en kemisk ljusproducerande enzymatisk reaktion. Varje gång en av nukleotiderna binder in till den komplementära DNA-strängen utsänds en ljussignal som påvisas, vilket gör att man kan utläsa sekvensen i ett pyrogram (till höger i bilden). ''Bild: Mia Brytting'']] | |
| − | '''Figur 20.''' ''Pyrosekvensering''. Metoden bygger på att en kemisk ljusproducerande enzymatisk reaktion. Varje gång en av nukleotiderna binder in till den komplementära DNA-strängen utsänds en ljussignal som påvisas, vilket gör att man kan utläsa sekvensen i ett pyrogram (till höger i bilden). ''Bild: Mia Brytting'' | ||
| − | |||
== Andra sekvenseringsmetoder == | == Andra sekvenseringsmetoder == | ||
| Rad 45: | Rad 39: | ||
För större mängder av DNA, såsom ett helt genom, är andra metoder som till exempel ”shotgun”-sekvensering mer lämplig. Vid denna metodik klyver man det DNA som skall sekvenseras i bitar och parallellsekvenserar alla fragmenten. Genom att sammanställa alla sekvenser med överlappande homologi kan långa sekvenssträngar fogas samman (se '''figur 21'''). | För större mängder av DNA, såsom ett helt genom, är andra metoder som till exempel ”shotgun”-sekvensering mer lämplig. Vid denna metodik klyver man det DNA som skall sekvenseras i bitar och parallellsekvenserar alla fragmenten. Genom att sammanställa alla sekvenser med överlappande homologi kan långa sekvenssträngar fogas samman (se '''figur 21'''). | ||
| − | + | [[Fil:molekylfig21.jpg|thumb|center|600px| '''Figur 21'''. Shotgun sekvensering bygger på sekvensering av flera fragment där sekvensinformationen sammanfogas med hjälp överlappning av sekvenshomologin. ''Bild: Mia Brytting'']] | |
| − | |||
| − | '''Figur 21'''. Shotgun sekvensering bygger på sekvensering av flera fragment där sekvensinformationen sammanfogas med hjälp överlappning av sekvenshomologin. ''Bild: Mia Brytting'' | ||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| + | == Massiv parallellsekvensering == | ||
| + | Genom att kombinera shotgun-tekniken och olika sekvenseringstekniker i stor skala har det idag blivit möjligt att sekvensera hela genom på kort tid. I dagsläget finns det flera olika system för massiv parallellsekvensering (''ref 5, 7-9)''. I detta kapitel beskrivs de tre som har kommit längst (år 2009) i sin utveckling. Dessa tekniker kallas ibland för ”''next generation sequencing''” | ||
| Rad 81: | Rad 64: | ||
| + | '''Figur 22.''' Sekvensering med 454-tekniken ''Bild: Roche, publicerad med företagets godkännande'' | ||
| + | [[Fil:molekyl22A.jpg|thumb|600px|center|||]] | ||
| − | + | [[Fil:molekyl22B.jpg|thumb|600px|center|||]] | |
| − | + | [[Fil:molekyl22C.jpg|thumb|600px|center|]] | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| + | [[Fil:molekyl22D.jpg|thumb|600px|center|||]] | ||
| + | ---- | ||
=== Solexa === | === Solexa === | ||
| Rad 106: | Rad 86: | ||
*7. Sevensering sker sedan med reversibel termineringssekvensering. Genom att tillsätta en mix av DNA-polymeras och de fyra nukleotiderna (alla med specifik fluorofor) kan sekvenseringen börja. Nukleotiderna har en stoppmolekyl vilket gör att endast en nukleotid kan inkorporeras per cykel. Med hjälp av laserkamera avläses vilken nukleotid (beroende på färg) samt i vilken region i kammaren som reaktion sker. Detta gör det möjligt att avläsa sekvensdata från flera olika templat på samma gång. Genom att ta bort stoppmolekylen och upprepa sekvenseringscykeln kan nästa nukleotid avläsas. Med denna teknik kan cirka 40*10<math>^6</math> sekvenser a’ 30-50 baser avläsas vilket innebär 1.6*10<math>^6</math> baser vid en sekvenseringsomgång. En hel analysomgång (exklusive databearbetningen) tar cirka tre dagar. | *7. Sevensering sker sedan med reversibel termineringssekvensering. Genom att tillsätta en mix av DNA-polymeras och de fyra nukleotiderna (alla med specifik fluorofor) kan sekvenseringen börja. Nukleotiderna har en stoppmolekyl vilket gör att endast en nukleotid kan inkorporeras per cykel. Med hjälp av laserkamera avläses vilken nukleotid (beroende på färg) samt i vilken region i kammaren som reaktion sker. Detta gör det möjligt att avläsa sekvensdata från flera olika templat på samma gång. Genom att ta bort stoppmolekylen och upprepa sekvenseringscykeln kan nästa nukleotid avläsas. Med denna teknik kan cirka 40*10<math>^6</math> sekvenser a’ 30-50 baser avläsas vilket innebär 1.6*10<math>^6</math> baser vid en sekvenseringsomgång. En hel analysomgång (exklusive databearbetningen) tar cirka tre dagar. | ||
| − | '''Figur 23.''' Sekvensering enligt solexatekniken. Bild: Illumina, publicerad med företagets godkännade. | + | |
| + | [[Fil:molekylfig23c.jpg|thumb|center|600px|| '''Figur 23.''' Sekvensering enligt solexatekniken. Bild: Illumina, publicerad med företagets godkännade.]] | ||
| + | |||
| + | ---- | ||
=== SOLiD === | === SOLiD === | ||
| Rad 126: | Rad 109: | ||
[[Fil:Cms 053243.jpg|thumb|center|900px|||'''Figur 24. '''. Sekvensering enligt SOLiD-teknik. Texten i figuren beskriver de olika stegen. ''Bild: Applied Biosystems en del av Life Technologies Group'', publicerad med företagets godkännande. | [[Fil:Cms 053243.jpg|thumb|center|900px|||'''Figur 24. '''. Sekvensering enligt SOLiD-teknik. Texten i figuren beskriver de olika stegen. ''Bild: Applied Biosystems en del av Life Technologies Group'', publicerad med företagets godkännande. | ||
| − | '''FÖR LÄSBAR BILD, KLICKA PÅ BILDEN, DU KOMMER DÅ TILL FILSIDAN DÄR DU ÅTER KAN KLICKA PÅ BILDEN TILL | + | '''FÖR LÄSBAR BILD, KLICKA PÅ BILDEN, DU KOMMER DÅ TILL FILSIDAN DÄR DU ÅTER KAN KLICKA PÅ BILDEN TILL LÄSBAR FÖRSTORINGSGRAD''']] |
== REFERENSER == | == REFERENSER == | ||
Nuvarande version från 12 april 2011 kl. 07.40
Till innehållsförteckningen för Referensmetodik:Molekylärbiologisk diagnostik
Sekvensering[redigera]
DNA-sekvensering är den process som används för att med biokemiska metoder bestämma ordningen av baserna adenin, guanin, cytosin och tymin i DNA. Sekvensen utgör den ärvda genetiska informationen och är därför viktig både i grundläggande forskning kring organismer, diagnostik, vid rättsmedicinska utredningar för att identifiera ärftliga sjukdomar och vid epidemiologiska utredningar. Den snabba utvecklingen inom området har möjliggjort storskaliga sekvenseringar av till exempel det mänskliga genomet. Med de nya sekvenseringsteknikerna får man längre och fler sekvenser med färre fel på kortare tid. Utvecklingen av enzymer, instrument och programvara pågår ständigt och de uppgifter på kapacitet man uppger idag är föråldrade inom kort.
Historik[redigera]
Det första virusgenomet (EBV) sekvenserades 1984, första bakteriegenomet, från Haemophilus influenzae 1995, jäst 1996 och första helgenomsekvensen från en människa 2007.
Under 1976-1977 utvecklade Maxam och Gilbert en DNA-sekvenseringsmetod baserad på kemisk modifikation av DNA och efterföljande delning vid specifika baser. Metoden benämns ibland som kemisk sekvensering. På grund av metodens komplexitet, omfattande användning av farliga kemikalier och svårigheter att använda den storskaligt användes denna metod endast under några år.
Sekvensering med Dideoxy-metoden[redigera]
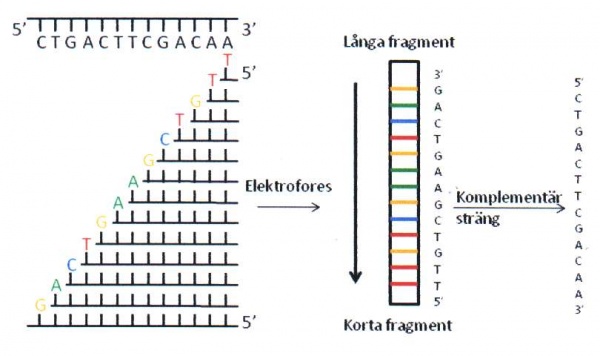
Under tidsperiod 1975-1977 utvecklade Sanger en sekvenseringsmetod för enkelsträngat DNA. Huvudprincipen bakom Sangers metod är användandet av dideoxy-nukleotider som orsakar stopp i DNA-syntesen, se figur 18. På 70- och 80-talen krävdes det att man framställde enkelsträngat DNA innan sekvensering med dideoxy-metoden kunde utföras. Oftast klonade man in det fragment man skulle sekvensera i en bakteriofag (M13) som har både en enkelsträngad och dubbelsträngad fas i sin replikationscykel. Man använde den dubbelsträngade DNA-formen för att klona in DNA-fragmentet. Sedan anrikade man den enkelsträngade formen för sekvenseringen. När PCR-metoden introducerades i slutet av 80-talet blev det möjligt att på ett enkelt sätt sekvensera med dideoxy-metoden. Med hjälp av PCR kan man amplifiera och analysera små mängder av templat. Med hjälp av färginmärkning av dideoxynukleotiderna separeras sekvenseringsprodukterna i kapillärer istället för stora gelelektroforessystem som tidigare användes (figur 19). Med all metodutveckling kring templatframställning, enzymer, färginmärkning och gel/kapillärseparering kan man idag läsa mellan 500-1000 långa fragment för upp till 384 prover samtidigt. Den ökande mängden sekvensdata har även krävt utveckling av dataprogrammen/mjukvaran för hantering av all sekvensinformation.
Pyrosekvensering[redigera]
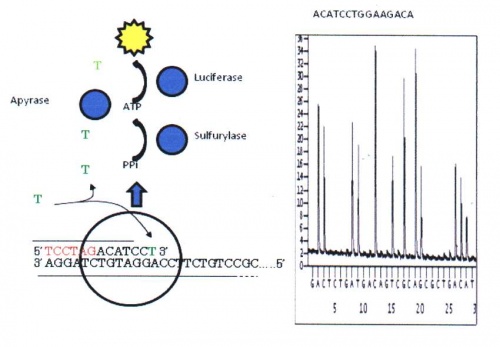
Tekniken utvecklades av en svensk grupp i slutet av 1990-talet. Metoden baseras på reaktioner där inbindandet av nukleotider till en komplementär DNA-sträng ger upphov till en ljusreaktion, se figur 20. Ljuset mäts via en fotometer. Ljusreaktionens intensitet är proportionerlig mot antalet inbundna nukleotider och eftersom endast "rätt" nukleotid kan binda in vid ett givet tillfälle kan man utläsa ordningsföljden. I början kunde man bara läsa 30-50 baser långa sekvenser. Idag har tekniken utvecklats så att 100 baser långa sekvenser kan utläsas. Denna metod används idag främst för att studera specifika mutationer eller variationer inom en begränsad region eller gen.
Andra sekvenseringsmetoder[redigera]
Det pågår en ständig utveckling av andra sekvenseringstekniker än de som nämnts ovan. Reversibel termineringssekvensering samt ligasbaserad sekvensering beskrivs under Solexa respektive SOLiD-teknikerna. En alternativ sekvenseringsmetod är att använda diskriminerande prober vid realtids-PCR, vid denna teknik kan endast en nukleotidposition per gång studeras sk SNP (single nucleotide polymorphisms) - analys. Ytterligare metoder finns och beskrivs i följande referenser (4-9).
Shotgun-sekvensering[redigera]
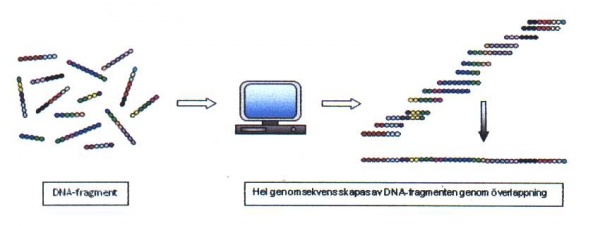
För större mängder av DNA, såsom ett helt genom, är andra metoder som till exempel ”shotgun”-sekvensering mer lämplig. Vid denna metodik klyver man det DNA som skall sekvenseras i bitar och parallellsekvenserar alla fragmenten. Genom att sammanställa alla sekvenser med överlappande homologi kan långa sekvenssträngar fogas samman (se figur 21).
Massiv parallellsekvensering[redigera]
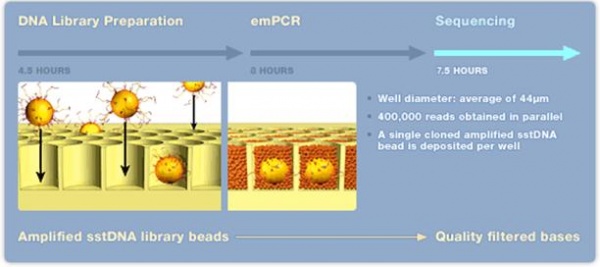
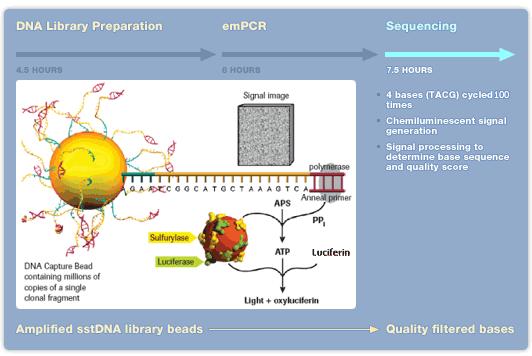
Genom att kombinera shotgun-tekniken och olika sekvenseringstekniker i stor skala har det idag blivit möjligt att sekvensera hela genom på kort tid. I dagsläget finns det flera olika system för massiv parallellsekvensering (ref 5, 7-9). I detta kapitel beskrivs de tre som har kommit längst (år 2009) i sin utveckling. Dessa tekniker kallas ibland för ”next generation sequencing”
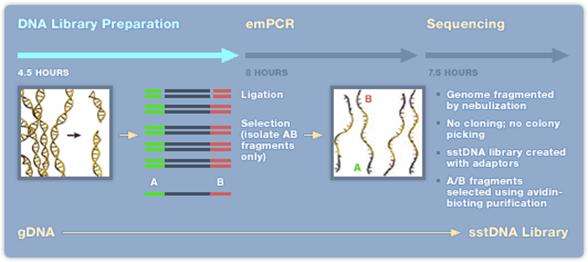
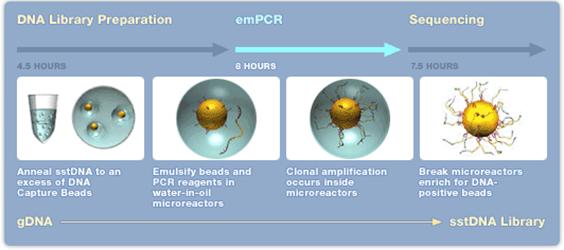
GS FLX Titanium - 454 teknologi[redigera]
- 1. Extraktion av DNA/RNA som skall sekvenseras.
- 2. Kemisk klyvning (nebulisering med kvävgas) av DNA/cDNA, behövs inte om det är korta (cirka 600-800 bp) PCR-fragment.
- 3. Ligering av korta DNA-strängar (adaptrar) A och/eller B (B adaptern är biotinylerad) till båda ändarna (5’ och 3’) av templat DNA.
- 4. DNA-fragment med B adaptern binds till en magnetisk kula med streptavidin. Man strävar efter att få ett fragment per kula. För att erhålla bra resultat krävs en separat titreringsomgång av templatet för att fastställa rätt förhållande mellan templat och kulor.
- 5. DNA-fragment som saknar B adaptern tvättas bort.
- 6. Magnetkulorna blandas i en vatten-oljeemulsion.
- 7. DNA-strängar med A och B adaptrar amplifieras via PCR-teknik i vatten-oljeemulsionen.
- 8. Emulsionen upplöses och kulorna placeras i en picotiterplatta, en kula per brunn. Till varje brunn tillsätts reaktionsmix för pyrosekvensering.
- 9. Pyrosekvenseringsreaktionen avläses med hjälp av en CCD kamera.
Efter 10-20 timmar (titreringssteget exkluderat) kan upp till 400 000 sekvenser om cirka 350-500 bp längd avläsas. Detta innebär cirka 400-600 millioner baser vid en sekvenseringsomgång. Uppgradering av denna teknik pågår och snart kommer det att vara möjligt att erhålla sekvenser av en längd kring 1000 bp. Idag tar hanteringen av all sekvenseringsdata längre tid än själva sekvenseringen.
Figur 22. Sekvensering med 454-tekniken Bild: Roche, publicerad med företagets godkännande
Solexa[redigera]
- 1. Extraktion av DNA/RNA som skall sekvenseras
- 2. Klyvning (sonikering alternativt nebulisering med kvävgas) av DNA/cDNA.
- 3. Ligering av korta DNA-strängar (adaptrar) A och/eller B till båda ändarna (5’ och 3’) av fragmenterat templat DNA.
- 4. Templatet binds till ytan i en flödeskammare med hjälp av hybridiseringsreaktioner (åtta olika prover kan analyseras samtidigt). I denna kammare finns redan ett stort antal av A och B adaptrarna bundna.
- 5. Genom homologi mellan adaptern på templatet och de som sitter i kammaren kommer nu adaptersekvenserna att binda till varandra (”så att det bildas en brygga (bridge)”). Detta tillåter att enskilda molekyler kan ampliferas via PCR innan sekvenseringsreaktionerna.
- 6. Amplifieringen av templaten sker i kammaren. De nybildade fragmenten binds in i kammaren i närheten av ursprungstemplatet. Detta kallas ibland för bridge-PCR. Detta gör att det finns flera små regioner i kammaren med amplifierat DNA.
- 7. Sevensering sker sedan med reversibel termineringssekvensering. Genom att tillsätta en mix av DNA-polymeras och de fyra nukleotiderna (alla med specifik fluorofor) kan sekvenseringen börja. Nukleotiderna har en stoppmolekyl vilket gör att endast en nukleotid kan inkorporeras per cykel. Med hjälp av laserkamera avläses vilken nukleotid (beroende på färg) samt i vilken region i kammaren som reaktion sker. Detta gör det möjligt att avläsa sekvensdata från flera olika templat på samma gång. Genom att ta bort stoppmolekylen och upprepa sekvenseringscykeln kan nästa nukleotid avläsas. Med denna teknik kan cirka 40*10 sekvenser a’ 30-50 baser avläsas vilket innebär 1.6*10 baser vid en sekvenseringsomgång. En hel analysomgång (exklusive databearbetningen) tar cirka tre dagar.
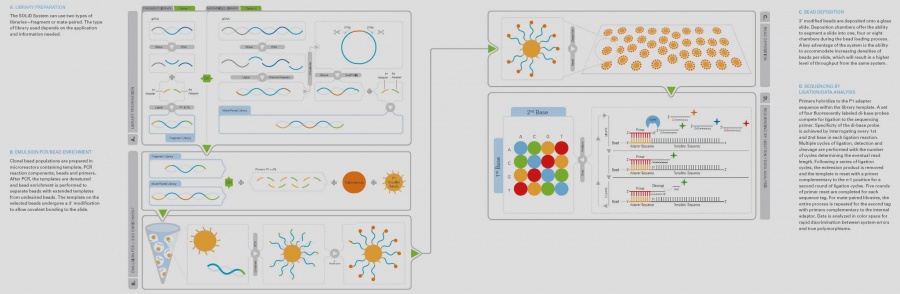
SOLiD[redigera]
- 1. Extraktion av DNA/RNA som skall sekvenseras.
- 2. Mekanisk klyvning (sonikering) av DNA/cDNA.
- 3. Separation av fragmenten via gelelektrofores. Fragment motsvarande en bestämd längd (ca 100 bp vid ett fragmentbibliotek och längre fragment vid t.ex genomisk kartläggning) skärs ut. Rening av fragmenten från gelen.
- 4. Korta DNA-strängar (adaptrar) A och B ligeras till båda ändarna (5’ och 3’) på templat DNA.
- 5. PCR-amplifiering av templatet med hjälp av primers riktade mot adapterregionerna. Genom att begränsa primerkoncentrationen för A-regionen och tillsätta magnetiska kulor som har den primern får man amplifiering av DNA från primer kopplade till magnetkulorna. Man försöker uppnå ett 1:1 förhållande mellan kulor och templatet.
- 6. PCR-amplifieringen sker i en vatten-oljeemulsion.
- 7. Emulsionen bryts när PCRen är färdig genom att butanol tillsätts och oljan tvättas bort.
- 8. Kulor med enkelsträngat DNA som har amplifierats från den magnetbundna primern anrikas. De tvättade kulorna med DNA blandas med en lösning med större polystyrenkulor vilka har en linker på ytan som kan binda B-adaptorn från DNA-kulan. Denna ”kulmix” läggs ovanpå en glycerollösning och centrifugeras. Efter centrifugering hamnar kulor utan DNA i en pellet och kulor som är bundna till polystyren kulan ligger kvar i en fas ovanpå glycerolen. Dessa kulor tvättas och DNA denatureras så det släpper från polystyrenkulan.
- 9. 3’ ändan av dessa strängar modifieras med terminal transferas så att de binder kovalent till glas. Kulorna deponeras på ett glas som kan delas in i ett, fyra eller åtta fält.
- 10. DNA sekvenseras med ligassekvensering. Ofta används 5 olika primers som binder olika långt ut från adaptorn. Proben som ligeras till primern har färgmolekylen på sig. Proben är en oktamer med enbart två unika baser, 256 olika prober per färg, totalt 1024 olika prober finns i probemixen (4). När proben bundit läses färgen av från varje kula i fyra olika kanaler. Efter detta klyvs 5’ änden bort på proben så nästa probe kan ligeras. När signal från sista proben detekterats rensas templatet och nästa primer kan binda in ett steg före föregående primers bindningsställe. Vid sekvensering av fragmentbibliotek läses 50 baser med start från adaptor. Fragmentet kan vara av olika längd men enbart 25-50 baser sekvenseras. Under våren 2009 kommer en uppgradering till SOLiD med möjlighet till läsning av 75 baser.
- 11. Att sekvensera ett fragmentbibliotek tar idag cirka 3-5 dagar. Man kan då erhålla cirka 100* baser vid en sekvenseringsomgång. Idag tar hanteringen av all sekvenseringsdata längst tid.
REFERENSER[redigera]
- 1 Maxam AM, Gilbert W. A new method for sequencing DNA. Proc. Natl. Acad. Sci. U.S.A. 1977;74:560-4.
- 2 Sanger F, Nicklen S, Coulson AR. DNA sequencing with chain-terminating inhibitors. Proceedings of the National Academy of Sciences USA 1977;74: 5463-5467.
- 3 Nyren P. Enzymatic method for continuous monitoring of DNA polymerase activity. Anal. Biochem. 1987;167:235-8.
- 4 Ahmadian A, Ehn M, Hober S. 2006. Pyrosequencing: History, biochemistry and future. Clinica. Chimica. Acta. 2006;363(1-2): 83-94.
- 5 Shendure JA, Porreca GJ, Church GM. Overview of DNA sequencing strategies. Current Protocols in Mol. Biol. 2008; 7.1.1-11.
- 6 Ausubel FM, Albright LM, Ju J. DNA sequencing. Current Protocols in Mol. Biol. 2008; 7.0.1-15.
- 7 Metzker ML. Emerging technologies in DNA sequencing. Genome Res. 2005;15(12):1767-76.
- 8 Ziebolz B, Droege M. Toward a new era in sequencing. Biotechnol Annu Rev. 2007;13:1-26.
- 9 Bentley DR. Whole-genome re-sequencing. Curr Opin Genet Dev. 2006;16(6):545-52.
- 10 Venter JC, Smith HO, Hood L. A new strategy for genome sequencing. Nature 1996; 381, 364-6.