Skillnad mellan versioner av "Bilaga 2. Bakteriologiska referensmaterial"
Hoppa till navigering
Hoppa till sök
| (13 mellanliggande sidversioner av samma användare visas inte) | |||
| Rad 2: | Rad 2: | ||
---- | ---- | ||
| − | |||
| − | + | == Bakteriologiska referensmaterial == | |
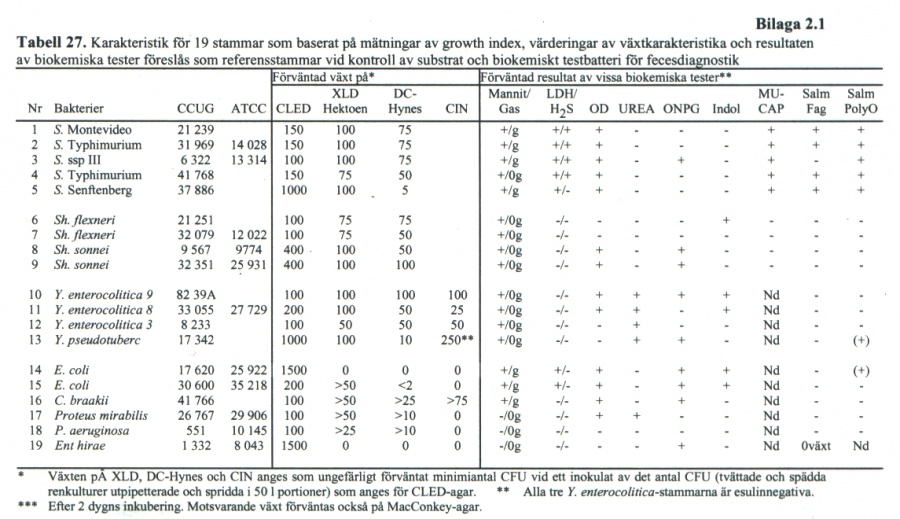
| − | + | [[Fil:Fecestabell27a3.jpg|thumb|900px|center||]] | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | + | ---- | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | + | === Referensstammar === | |
| − | Referensstammar | ||
| − | |||
| − | |||
| − | |||
| − | |||
| − | För | + | *För serotypning |
| − | + | ** O4 -''S''. Typhimurium---- CCUG 31 969 | |
| + | ** O7 -''S''. Virchow---- CCUG 12 653 | ||
| + | ** O8 -''S''. Newport---- CCUG 21283 | ||
| + | ** O9 -''S.'' Enteritidis---- CCUG 34136 | ||
| + | ** O10 –''S.'' Anatum CCUG ---- 37885 | ||
| + | ** O19 –''S.'' Senftenberg---- CCUG 37 886 | ||
| − | + | '''''Campylobacter''''' | |
| − | |||
| − | |||
| − | + | *Referensstammar för substratkontroll vid varje gjutning: | |
| − | + | **''C. jejuni'' CCUG 11284A god växt | |
| − | + | **''E. coli'' CCUG 17620 hämmad helt eller delvis | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | + | *Vid lotkontroll dessutom: | |
| + | **''C. coli'' CCUG 11283 god växt | ||
| − | + | *För kontroll av odlingsförfarandet används | |
| − | + | **''C. jejuni'' CCUG 11284A | |
| − | |||
| − | + | *För identifiering dessutom | |
| − | + | **''C. lari'' CCUG 23947 | |
| − | + | **''C. upsaliensis'' CCUG 19559 | |
| − | |||
| − | |||
| − | |||
| − | + | '''Tarmpatogena ''E. coli''''' | |
| − | |||
| − | + | Målgener för PCR-screening | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | + | Negativ kontroll för samtliga PCR, ''E. coli'' ATCC 11775 | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | Vibrio cholerae | + | '''''Vibrio cholerae''''' |
| − | |||
| − | |||
| − | + | *''Vibrio cholerae'' O-1 CCUG 9119 | |
| − | + | *''Vibrio cholerae'' O-139 CCUG 34708 | |
| − | + | '''''Vibrionaceae''''' | |
| − | |||
| − | |||
| − | + | *''Aeromonas hydrophila'' CCUG 217 | |
| + | *''Plesiomonas shigelloides'' CCUG 27761 | ||
| + | *''Vibrio parahaemolyticus'' CCUG 51447 | ||
| − | |||
| − | + | '''''Helicobacter pylori''''' | |
| − | + | *''H. pylori'' NCTC 11637 (CCUG 17874) | |
| − | |||
| − | |||
| − | |||
| − | + | '''''Clostridium difficile''''' | |
| − | |||
| − | + | ''C. difficile'' CCUG 4938T används för kontroll av CCFA-plattor, som jämförelse vid luppmikroskopi, som kontroll på den anaeroba miljön och som kontrollstam vid eventuell resistensbestämning | |
| − | |||
| − | |||
| − | + | '''Kontaminantstammar''' att använda som referensstammar vid lottestning och fortlöpande tillverkningskontroll av fecessubstrat enligt referensmetodiken. | |
| − | |||
| − | |||
| − | |||
| − | + | '''Matförgiftningsstammar''' | |
| − | + | *''Bacillus cereus'' CCUG 10 781 | |
| − | + | *''Clostridium perfringens'' CCUG 1795 | |
| + | *''Staphylococcus aureus ''CCUG 17 621 | ||
| − | + | === Referenssera === | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | + | *''Salmonella'' | |
| − | + | **Polyvalent O-antiserum (Reagensia AB) | |
| − | + | **Polyvalent H-antiserum (Reagensia AB) | |
| − | + | **Faktorantiserum (Reagensia AB) | |
| − | |||
| − | |||
| − | |||
| − | + | *''Shigella'' | |
| − | + | **''Sh. dysenteriae'' 1-13 (Reagensia AB) | |
| + | **''Sh. flexneri'' (Reagensia AB) | ||
| + | **''Sh. flexneri'' monoklonaler (Reagensia AB) | ||
| + | **''Sh. boydii'' 1-18 (Reagensia AB) | ||
| + | **''Sh. sonnei'' SoR (Reagensia AB) | ||
| − | + | *''Yersinia'' | |
| − | + | **''Yersinia'' O3 och O9 (Reagensia AB) | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | + | *''Vibrio cholerae'' | |
| − | + | **''Vibrio cholerae'' Polyvalent O-1 (Denka Seiken) | |
| + | **''Vibrio cholerae'' O-139 (Denka Seiken) | ||
| − | + | *EHEC | |
| − | + | **''Escherichia coli'' O-157 (Denka Seiken) | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
[[Kategori:Laboratoriediagnostik-Infektioner i mage och tarm ]] | [[Kategori:Laboratoriediagnostik-Infektioner i mage och tarm ]] | ||
Nuvarande version från 19 mars 2013 kl. 15.41
Till innehållsförteckningen för Referensmetodik: Tarminfektioner, 2:a upplagan 2002
Bakteriologiska referensmaterial[redigera]
Referensstammar[redigera]
- För serotypning
- O4 -S. Typhimurium---- CCUG 31 969
- O7 -S. Virchow---- CCUG 12 653
- O8 -S. Newport---- CCUG 21283
- O9 -S. Enteritidis---- CCUG 34136
- O10 –S. Anatum CCUG ---- 37885
- O19 –S. Senftenberg---- CCUG 37 886
Campylobacter
- Referensstammar för substratkontroll vid varje gjutning:
- C. jejuni CCUG 11284A god växt
- E. coli CCUG 17620 hämmad helt eller delvis
- Vid lotkontroll dessutom:
- C. coli CCUG 11283 god växt
- För kontroll av odlingsförfarandet används
- C. jejuni CCUG 11284A
- För identifiering dessutom
- C. lari CCUG 23947
- C. upsaliensis CCUG 19559
Tarmpatogena E. coli
Målgener för PCR-screening
Negativ kontroll för samtliga PCR, E. coli ATCC 11775
Vibrio cholerae
- Vibrio cholerae O-1 CCUG 9119
- Vibrio cholerae O-139 CCUG 34708
Vibrionaceae
- Aeromonas hydrophila CCUG 217
- Plesiomonas shigelloides CCUG 27761
- Vibrio parahaemolyticus CCUG 51447
Helicobacter pylori
- H. pylori NCTC 11637 (CCUG 17874)
Clostridium difficile
C. difficile CCUG 4938T används för kontroll av CCFA-plattor, som jämförelse vid luppmikroskopi, som kontroll på den anaeroba miljön och som kontrollstam vid eventuell resistensbestämning
Kontaminantstammar att använda som referensstammar vid lottestning och fortlöpande tillverkningskontroll av fecessubstrat enligt referensmetodiken.
Matförgiftningsstammar
- Bacillus cereus CCUG 10 781
- Clostridium perfringens CCUG 1795
- Staphylococcus aureus CCUG 17 621
Referenssera[redigera]
- Salmonella
- Polyvalent O-antiserum (Reagensia AB)
- Polyvalent H-antiserum (Reagensia AB)
- Faktorantiserum (Reagensia AB)
- Shigella
- Sh. dysenteriae 1-13 (Reagensia AB)
- Sh. flexneri (Reagensia AB)
- Sh. flexneri monoklonaler (Reagensia AB)
- Sh. boydii 1-18 (Reagensia AB)
- Sh. sonnei SoR (Reagensia AB)
- Yersinia
- Yersinia O3 och O9 (Reagensia AB)
- Vibrio cholerae
- Vibrio cholerae Polyvalent O-1 (Denka Seiken)
- Vibrio cholerae O-139 (Denka Seiken)
- EHEC
- Escherichia coli O-157 (Denka Seiken)