Skillnad mellan versioner av "Genotypisk MRSA-verifiering"
| (5 mellanliggande sidversioner av samma användare visas inte) | |||
| Rad 10: | Rad 10: | ||
===Indikation=== | ===Indikation=== | ||
| − | Verifiering av | + | Verifiering av MRSA genom att påvisa ''mecA''- och ''nuc''-gen. Metoden är avsedd att användas för verifiering av misstänkta kolonier av MRSA på agarplatta som identifierats i rutin-eller screeningprover. För presumptivt påvisande av MRSA i screeningprover hänvisas till artikel [[MRSA-screening enligt Halmstadmodellen]]. |
| − | + | Genen ''mecA'' kodar för ett penicillinbindande protein (PBP2a) med sänkt affinitet för betalaktamantibiotika,vilket gör olika arter av ''mecA''-bärande stafylokocker resistenta mot alla betalaktam-antibiotika inkluderande betalaktamasstabila isoxazolylpenicilliner. Särskilt allvarligt är meticillinresistens hos ''S. aureus'', eftersom antibiotika ur denna grupp sedan länge utgjort standardterapi vid infektioner med sådana bakterier. | |
| − | + | Metoden som beskrivs här är utformad för att påvisa förekomst av ''mecA''-gen hos stafylokocker genom PCR-amplifiering av målområdet och detektion med SYBR Green med realtids-PCR-instrument. I kombination med påvisande av ''nuc''-gen utförd med samma DNA-preparation är den avsedd för verifiering av fynd av MRSA. Primrar för ''mecA''-amplifiering påvisar också en variant av ''mecA''-genen som påvisats bland annat i ''S. aureus''-stammen LGA251 (1, 2) | |
| − | + | === Material=== | |
| − | Utgångsmaterialet är | + | Utgångsmaterialet är renkultur på blodagar av misstänkta bakterier. För amplifiering används PCR-instrument LightCycler 2.0 (Roche Diagnostics) och reagenser LightCycler FastStart DNA MasterPLUS SYBR Green I (Roche Diagnostics). |
| − | Referensstammar: | + | *Referensstammar: |
| − | + | ** CCUG 35603 ''Staphylococcus aureus'' ''mecA''+ | |
| − | + | ** CCUG 17621 ''Staphylococcus aureus'' ''mecA''- | |
| − | + | ** CCUG 63582 ''Staphylococcus aureus'' med ''mecA''<sub>LGA251</sub> | |
| − | + | ||
| − | + | *Primrar | |
| − | + | **PCR primer MEC9 5´- TCACCAGGTTCAAC[Y]CAAAA (1) | |
| − | + | ** PCR primer MEC10 5´- CCTGAATC[W]GCTAATAATATTTC (1) | |
| − | + | ** PCR primer NUC4 5’-TCAAGTCTAAGTAGCTCAGCAAATGC (3) | |
| − | + | ** PCR primer NUC5 5’-GAAGTTGCACTATATACTGTTGGATCT TC (3) | |
| − | + | ||
| + | ===Analys=== | ||
| − | + | ====DNA-frisättning==== | |
| − | Slamma en liten koloni eller motsvarande mängd bakterier i 100 | + | Slamma en liten koloni eller motsvarande mängd bakterier i 100 µL sterilt avjoniserat vatten i ett sterilt 1.5 mL eppendorfrör. Se till att bakterierna blir ordentligt uppslammade. |
| + | |||
| + | Mängden bakterier är lagom för analys när man kan ana en lätt grumlighet. | ||
| − | PCR | + | ====PCR==== |
| − | Varje isolat analyseras i två separata 20 µL PCR-reaktioner, en för detektion av nuc och en för | + | Varje isolat analyseras i två separata 20 µL PCR-reaktioner, en för detektion av ''nuc'' och en för ''mec''A. PCR-reaktionerna innehåller PCR-reagens enligt tillverkarens rekommendation, 0.5 µM av respektive primrar (NUC4/NUC5 respektive MEC9/MEC10) och 2 µL templat (suspension av bakterie). |
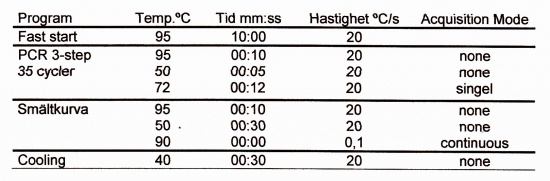
Följande amplifieringsprofil används: | Följande amplifieringsprofil används: | ||
| + | [[Fil:MRSAPCR.jpg|thumb|550px|center||]] | ||
| + | |||
| + | ====Tolkning av resultatet==== | ||
| − | + | Bestämning av smältpunkt (Tm) för PCR-produkter från ''nuc'' respektive ''mecA'' amplifieringsprodukter är avgörande för tolkning av resultat. Förväntade Tm för ''mecA'' PCR-produkt är runt 78,6°C, för ''mecA''<sub>LGA251</sub> runt 80.0°C och för ''nuc'' runt 79,2°C. | |
| − | + | ||
| − | + | ==REFERENSER== | |
| − | |||
| − | |||
| − | |||
| − | 1. Laura García-Álvarez et al. Meticillin-resistant Staphylococcus aureus with a novel mecA homologue in human and bovine populations in the UK and Denmark: a descriptive study. Lancet Infect Dis 2011;11: 595–603 | + | *1. Laura García-Álvarez et al. Meticillin-resistant Staphylococcus aureus with a novel mecA homologue in human and bovine populations in the UK and Denmark: a descriptive study. Lancet Infect Dis 2011;11: 595–603 |
| − | 2 Shore AC et al. Detection of staphylococcal cassette chromosome mec type XI carrying highly divergent mecA, mecI, mecR1, blaZ, and ccr genes in human clinical isolates of clonal complex 130 methicillin-resistant Staphylococcus aureus. Antimicrob Agents Chemother. 2011 Aug;55(8):3765-73. Epub 2011 Jun 2 | + | *2 Shore AC et al. Detection of staphylococcal cassette chromosome mec type XI carrying highly divergent mecA, mecI, mecR1, blaZ, and ccr genes in human clinical isolates of clonal complex 130 methicillin-resistant Staphylococcus aureus. Antimicrob Agents Chemother. 2011 Aug;55(8):3765-73. Epub 2011 Jun 2 |
| − | 3 Peter Nilsson, Klinisk mikrobiologi, Hallands sjukhus Halmstad. Egen design. | + | *3 Peter Nilsson, Klinisk mikrobiologi, Hallands sjukhus Halmstad. Egen design. |
[[Kategori:Smittskyddslagens sjukdomar]] | [[Kategori:Smittskyddslagens sjukdomar]] | ||
Nuvarande version från 24 januari 2013 kl. 13.38
Artikel publicerad april 2012. Innehåll preliminärt i väntan på konsensusförfarande
Till innehållsförteckningen för Referensmetodik:Smittskyddslagens sjukdomar
och
förgreningssidan MRSA
Genotypisk MRSA-verifiering exemplifierad med metod för LightCycler[redigera]
Indikation[redigera]
Verifiering av MRSA genom att påvisa mecA- och nuc-gen. Metoden är avsedd att användas för verifiering av misstänkta kolonier av MRSA på agarplatta som identifierats i rutin-eller screeningprover. För presumptivt påvisande av MRSA i screeningprover hänvisas till artikel MRSA-screening enligt Halmstadmodellen.
Genen mecA kodar för ett penicillinbindande protein (PBP2a) med sänkt affinitet för betalaktamantibiotika,vilket gör olika arter av mecA-bärande stafylokocker resistenta mot alla betalaktam-antibiotika inkluderande betalaktamasstabila isoxazolylpenicilliner. Särskilt allvarligt är meticillinresistens hos S. aureus, eftersom antibiotika ur denna grupp sedan länge utgjort standardterapi vid infektioner med sådana bakterier.
Metoden som beskrivs här är utformad för att påvisa förekomst av mecA-gen hos stafylokocker genom PCR-amplifiering av målområdet och detektion med SYBR Green med realtids-PCR-instrument. I kombination med påvisande av nuc-gen utförd med samma DNA-preparation är den avsedd för verifiering av fynd av MRSA. Primrar för mecA-amplifiering påvisar också en variant av mecA-genen som påvisats bland annat i S. aureus-stammen LGA251 (1, 2)
Material[redigera]
Utgångsmaterialet är renkultur på blodagar av misstänkta bakterier. För amplifiering används PCR-instrument LightCycler 2.0 (Roche Diagnostics) och reagenser LightCycler FastStart DNA MasterPLUS SYBR Green I (Roche Diagnostics).
- Referensstammar:
- CCUG 35603 Staphylococcus aureus mecA+
- CCUG 17621 Staphylococcus aureus mecA-
- CCUG 63582 Staphylococcus aureus med mecALGA251
- Primrar
- PCR primer MEC9 5´- TCACCAGGTTCAAC[Y]CAAAA (1)
- PCR primer MEC10 5´- CCTGAATC[W]GCTAATAATATTTC (1)
- PCR primer NUC4 5’-TCAAGTCTAAGTAGCTCAGCAAATGC (3)
- PCR primer NUC5 5’-GAAGTTGCACTATATACTGTTGGATCT TC (3)
Analys[redigera]
DNA-frisättning[redigera]
Slamma en liten koloni eller motsvarande mängd bakterier i 100 µL sterilt avjoniserat vatten i ett sterilt 1.5 mL eppendorfrör. Se till att bakterierna blir ordentligt uppslammade.
Mängden bakterier är lagom för analys när man kan ana en lätt grumlighet.
PCR[redigera]
Varje isolat analyseras i två separata 20 µL PCR-reaktioner, en för detektion av nuc och en för mecA. PCR-reaktionerna innehåller PCR-reagens enligt tillverkarens rekommendation, 0.5 µM av respektive primrar (NUC4/NUC5 respektive MEC9/MEC10) och 2 µL templat (suspension av bakterie).
Följande amplifieringsprofil används:
Tolkning av resultatet[redigera]
Bestämning av smältpunkt (Tm) för PCR-produkter från nuc respektive mecA amplifieringsprodukter är avgörande för tolkning av resultat. Förväntade Tm för mecA PCR-produkt är runt 78,6°C, för mecALGA251 runt 80.0°C och för nuc runt 79,2°C.
REFERENSER[redigera]
- 1. Laura García-Álvarez et al. Meticillin-resistant Staphylococcus aureus with a novel mecA homologue in human and bovine populations in the UK and Denmark: a descriptive study. Lancet Infect Dis 2011;11: 595–603
- 2 Shore AC et al. Detection of staphylococcal cassette chromosome mec type XI carrying highly divergent mecA, mecI, mecR1, blaZ, and ccr genes in human clinical isolates of clonal complex 130 methicillin-resistant Staphylococcus aureus. Antimicrob Agents Chemother. 2011 Aug;55(8):3765-73. Epub 2011 Jun 2
- 3 Peter Nilsson, Klinisk mikrobiologi, Hallands sjukhus Halmstad. Egen design.