Strand Displacement Amplification (SDA)
Till innehållsförteckningen för Referensmetodik:Molekylärbiologisk diagnostik
Strand Displacement Amplification (SDA)
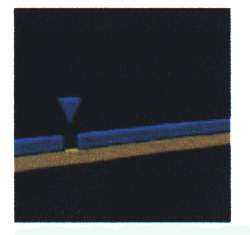
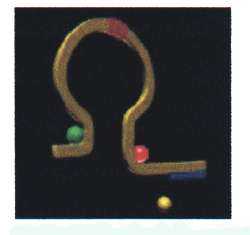
SDA-tekniken är en målbaserad isotermal amplifieringsmetod och ett alternativ till PCR i ett kommersiellt system (figur16). SDA-metoden används framförallt för detektion av Chlamydia trachomatis och Neisseria gonorrhoeae, men den används även för specifik detektion av Mycoplasma pneumoniae, Chlamydophila pneumoniae, Legionella pneumophila och Mycobacterium tuberculosis i samma system. Denaturering sker endast en gång i början av processen. Metoden är enzymatisk vilket gör att inga temperaturväxlingar behövs och att hela processen kan utföras vid 52 °C. SDA kan beskrivas med tre delmoment; generering av mål-DNA kopia, exponentiell SDA-amplifiering och detektion. Metoden baseras på förmågan hos DNA-polymeraset att initiera DNA-replikation (kopiering) vid ett brott i enkelsträngat DNA, samtidigt som det ersätter nukleotider i den uppbrutna strängen (eng. nick translation). Den ersatta, nya, enkelsträngen av DNA används sedan som substrat då reaktionen fortsätter med primerinbindning, klyvning och replikation. Öppningen (brottet) i den enkelsträngade DNA-molekylen produceras av ytterligare ett enzym, ett restriktionsendonukleas.
De startsekvenser, primers, som används i reaktionen har en målspecifik region på 15-20 baspar. Med hjälp av två primerpar skapas först en kopia av mål-DNA sekvensen. Då det ena primerparet innehåller en specifik sekvens som känns igen av restriktionsenzymet BsoB I, kommer den nya DNA-kopian att innehålla klyvningsområdet för BsoB I. Genom att använda modifierade cytosin (dCTPS) under amplifieringsprocessen hindras restriktionsenzymet från att klippa dubbelsträngat. Restriktionsenzymet introducerar en enkelsträngad öppning från vilken polymeraset initierar DNA-replikering och skapar en kopia. Eftersom polymeraset saknar exonukleasaktivitet ”knuffas” framförliggande DNA bort som en hel DNA-sträng (ny kopia). Restriktionsenzymet kan återigen klyva och polymeraset kopierar om och om igen. Varje ny kopia kan agera som templat för vidare hybridisering, elongering och restriktionsklyvning.
Ersättning av den öppnade strängen sker i båda riktningar vilket ger en exponentiell amplifiering av målsekvensen. De amplifierade produkterna detekteras med hjälp av Fluorescence Resonance Energy Transfer (FRET), där en fluorescerande detektorprobe inmärkt med två olika fluorescerande molekyler, ”reporter” och ”quencher”, används. Proben har en ”hårnålsstruktur” och den binder till en specifik del av mål-DNA-sekvensen. Proben har även den sekvens som känns igen av restriktionsenzymet. Polymeraset förlänger proben som därefter kopieras och rätas ut till dubbelsträngat DNA. Då proben klyvs separeras de två fluoroforerna, vilket gör att fluorescensen från reportern inte längre släcks ut, utan maximeras. Ökningen av fluorescens i provet detekteras i en fluorescensläsare och analyseras.
Figur 16 (1-8). Strand Displacement Amplification Bild: Copyright Becton Dickinson, http://www.bd.com