Vankomycinresistenta enterokocker (VRE), referensmetodik
Till innehållsförteckningen för
Vankomycinresistenta enterokocker (VRE), referensmetodik
Påvisning av enterokocker i mikrobiologisk rutindiagnostik
Primär diagnostik av VRE förutsätter fenotypisk artdiagnostik av Enterococcus faecalis och Enterococcus faecium, och att man säkert kan skilja dessa arter från övriga enterokocker och från andra naturligt vankomycinresistenta grampositiva bakterier som Lactobacillus, Lactococcus och Pediococcus spp.
Minimikriterier för att artbestämma enterokocker är: typisk kolonimorfologi samt positiva tester för pyrrolydonylarylamidas (PYR) och galla- eskulin (1). Utöver detta är enterokocker positiva i agglutinationstest för Lancefields grupp-D-antigen (observera att även grupp-D-streptokocker är positiva i galla-eskulin, men negativa i PYR).
För att skilja de olika enterokockarterna från varandra används jäsning av arabinos (E. faecalis är negativ medan övriga enterokocker är positiva) alternativt tellurit (E. faecalis är positiv och E. faecium är negativ), rörlighet (både E. faecalis och E. faecium är orörliga) samt känslighet för penicillin och cefadroxil. E. faecalis och E. faecium har högre grad av resistens mot dessa betalaktamer än E. casseliflavus och E. gallinarum. De två sistnämnda arterna isoleras dock sällan från kliniska prover, men de utgör ett problem i samband med screening av feces- eller perianalprover på förekomst av glykopeptidresistenta enterokocker.Om man använder screeningmetoder med medier som innehåller selekterande och differentierande substanser, inklusive vankomycin, medför det att även andra bakterier (Lactobacillus, Lactococcus och Pediococcus spp)) som alltid är resistenta mot vankomycin och/eller teikoplanin fastnar i screenodlingen. Genom att först göra gramfärgning och PYR-test kan icke relevanta arter sorteras bort.
Diagnostiska minimikriterier för primär artdiagnostik av enterokocker, se bilaga 2.
Påvisning av enterokocker med nedsatt känslighet för vankomycin
Referensgruppen för antibiotikafrågor – metodgruppen (RAF-M, från 2011 Nordicast (Ref)) rekommenderar lappdiffusionsmetoden för att upptäcka vankomycinresistens (för zonbrytpunkter och detaljerad metod se www.srga.org.). Vid minskad hämningszon och/eller oskarp zonkant med gradvis avtagande växt runt vankomycinlappen ska isolatet karakteriseras vidare genom molekylärbiologisk påvisning av resistensgen (se nedan).
Lappdiffusionsmetoden är beroende av korrekt inokulat samt av att avläsaren är uppmärksam på zonkantens kvalitet. Om förhållandena inte är optimala kan enstaka VRE-isolat missas i denna test varför kvalitetssäkring av diagnostiken genom att delta i externa kvalitetskontrollprogram är av stor vikt.
Art- och resistensbestämning med automatiserade system
För art- och resistensbestämning är automatiserade system, som exempelvis Vitek 2 (bioMerieux) och Phoenix 100 (Becton Dickinson), enligt litteraturen väl anpassade för att upptäcka vankomycinresistens. Vissa problem finns dock rapporterade vad gäller artidentifiering av Enterococcus spp (2, 3, 4) och fyndet bör därför verifieras med referensmetod enligt ovan. Liksom vid manuell metodik ska även isolat som påvisas med automatiserade system verifieras med genetiskt påvisande av resistensgen.
Verifiering av VRE med molekylärbiologisk metod (PCR)
Isolat som har identifierats som VRE med fenotypiska metoder bör verifieras med molekylärbiologisk metod, PCR (Polymerase chain reaction), där ligas-generna som medierar vankomycinresistens (i första hand vanA och vanB) påvisas. Flera metoder för detta med konventionell PCR eller realtids-PCR, har publicerats och är kommersiellt tillgängliga.
Metod för genetisk verifiering av art kan utföras med PCR för artspecifika ligas- gener (ddl-E. faecium/faecalis/casseliflavus/gallinarum). För referenser samt beskrivning av en PCR-metod för att verifiera VRE, se bilaga 4 och rapportens nästa avsnitt.
Sammanfattning av avsnitt 1
- För korrekt detektion av VRE bör enterokocker artbestämmas och samtliga relevanta fynd resistensbestämmas med lappdiffusionstest för vankomycin.
- Vid lappdiffusionsmetod är inokulatets tjocklek samt zonkantens karaktär av största vikt.
- Vid minskad hämningszon och/eller oskarp zonkant med gradvis avtagande växt runt vankomycinlappen ska isolatet karakteriseras vidare genom genetisk påvisning av resistensgen.
Metod för screening inklusive genetisk verifiering av VRE
Detta avsnitt beskriver metod för screening och genetisk verifiering av generna vanA och vanB hos E. faecalis och E. faecium. Metoden består av anrikning av provet över natt i en selektiv buljong med vankomycin, följt av analys av buljongen med hjälp av realtids-PCR för att detektera resistensgenerna vanA och vanB. Med denna metod kan negativa prov besvaras relativt snabbt. Prov som är positiva i PCR-analysen måste odlas ut för att möjliggöra identifiering av enterokockerna.
Anrikning i selektiv buljong
Vid screening av VRE bör provet anrikas i selektiv buljong eftersom det ger bättre utbyte än vid direkt odling på selektiv agarplatta (5). Basen kan utgöras av Todd- Hewitt-buljong eller Galla-eskulin-buljong (6). För selektionen tillsätts vankomycin 4 mg/L och aztreonam 60 mg/L (protokoll, se bilaga 3). Koncentrationen av vankomycin bör vara 4 mg/L eftersom också låggradig resistens kan orsakas av vanB-genen. För vildtypspopulationen av E. faecium och E. faecalis dominerar MIC-värden 0,5–4 mg/L, medan MIC-värdena kan variera 4–1024 mg/L om bakterien har vanB-genen (EUCAST).
Vid provsättningen bryts provtagningspinnen ned i buljongen varefter buljongerna inkuberas i skakinkubator 35°C 16–24 h. Efter inkubation analyseras alla buljonger. Enterokocker bör ge ett svart färgomslag i Galla-eskulinbuljongen, men även ljusa buljonger kan innehålla stora mängder VRE. Detektionen för vanA- och vanB-generna sker med hjälp av PCR direkt på buljongen eller på bakteriekolonier efter utodling på selektivt substrat.
Detektion med realtids-PCR för vanA och vanB
Extraktion av DNA från buljongkulturerna utförs bäst i något av de automatiseradesystem som finns på marknaden.PCR-metoden som beskrivs i bilaga 4 är en modifierad duplex in-house-PCR (8) som har utvecklats vid Klinisk Mikrobiologi i Halmstad. Den är utvärderad för FRET-hybridiseringsprober i LightCycler (Roche) och TaqMan-prober i Rotor- Gene 6000. Sekvenserna för vanA och vanB ligger i konserverade regioner och detekterar varianterna vanB1, vanB2 och vanB3 (7,8,9) (primers, prober och temperaturprofil, se bilaga 4). Alternativa egenutvecklade eller kommersiella metoder för att genetiskt verifiera vanA och vanB med validerade prestanda kan ersätta den här beskrivna PCR-metoden.
Vid positivt PCR-resultat odlas buljongen ut, och misstänkta enterokocker verifieras med art- och resistensbestämning enligt ovan. Observera att van-generna kan förekomma även hos bakterier i den normala tarmfloran, till exempel är klostridier och alla positiva PCR-fynd måste verifieras som VRE.
Detektion med utodling
Anrikningsbuljongerna kan odlas ut på selektivt kromogent substrat med vankomycin, alternativt icke-selektivt medium med vankomycinlapp (7).
Plattorna inkuberas i 24 h för att ge lämplig kolonistorlek att arbeta vidare med. Misstänkta kolonier verifieras med fenotypiska metoder för art- och resistensbestämning, och van-gen verifieras med PCR.
Artbestämning av VRE med PCR för ddlE.
Som komplement till artbestämningen med biokemiska metoder kan en PCR för artspecifika ligas-gener (ddlE) användas. PCR-metoden skiljer på E. faecalis och E. faecium. Kommersiella eller egenutvecklade metoder kan användas för detta. I litteraturen beskrivs metoder för detta som block-PCR med detektion på gel (5). Samma protokoll har även utvecklats för realtidsplattform, se bilaga 4.
Sammanfattning av avsnitt 3
- Anrikning bör för acceptabel känslighet ske i selektiv buljong. Koncentrationen av vankomycin bör vara 4 mg/L.
- För att få snabbt utsvar av negativa prov bör buljongerna analyseras med realtids-PCR för vanA och vanB. Misstänkta enterokocker måste artbestämmas och resistensbestämmas samt genomgå genetisk verifiering av van-typ.
Epidemiologiska typningsmetoder för VRE
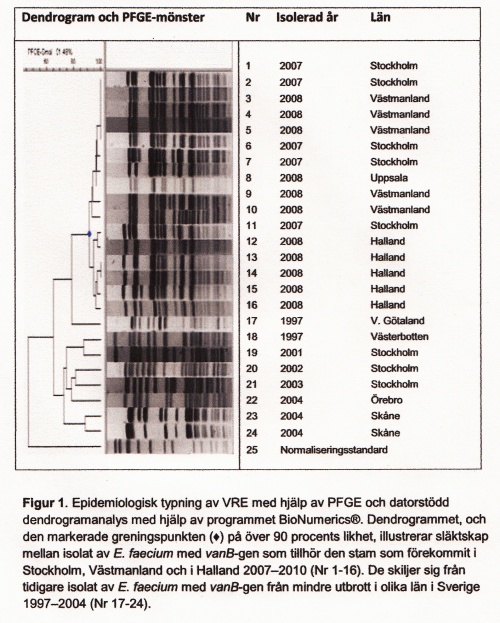
Epidemiologisk typning av bakterier används i första hand för att jämföra bakterieisolat då misstanke om smittspridning finns. För att identifiera en sannolik smittkedja krävs i första hand ett epidemiologiskt samband i tid och rum mellan patienter/personer, och dessutom ett samband mellan olika isolat som visats genom överensstämmande typningsresultat. Rekommenderad metodik för epidemiologisk typning av enterokocker, liksom för de flesta bakteriearter är pulsfältsgelelektrofores (PFGE), en metod som baserar sig på klyvning av bakteriens DNA med restriktionsenzymer följt av separation av 17 DNA-fragment i en agarosgel, och analys av bandmönster (Figur 1). För varje bakterieart måste testbetingelserna optimeras, både i val av restriktionsenzym och i övriga förhållanden i testsystemet. I den mån det finns internationellt accepterade metoder är det fördelaktigt att följa dessa, men sådana finns tyvärr endast för ett fåtal bakteriearter som till exempel Staphylococcus aureus, särskilt meticillinresistenta S. aureus (MRSA).
Internationellt accepterade kriterier för att bedöma samband mellan isolat kan ofta användas vid analys av isolatens bandmönster, men frågeställningen i det enskilda fallet, samt erfarenhet av den lokala eller nationella situationen, måste också vägas in vid bedömning. En av nackdelarna med PFGE är att resultaten inte är numeriska och därmed inte enkelt kommunicerbara mellan laboratorier. Trots detta har PFGE använts och används som en av de mest diskriminerande typningsmetoderna för MRSA, PNSP och för bakterier som tillhör familjen Enterobacteriaceae och metoden är således väl etablerad. Den kommer under överskådlig tid att finnas tillgänglig vid referenslaboratorium för att analysera misstänkta anhopningar av bakterieisolat och för nationell övervakning.
Det finns alternativa DNA-baserade typningsmetoder för enterokocker som till exempel MLST (Multi Locus Sequence Typing) och MLVA (Multi Locus Variable Number of Tandem Repeats Analysis). MLST baseras på sekvensering av sju ”house-keeping genes” vilket gör den dyrbar och mindre snabb, men fördelen med metoden är att resultatet lätt kan kommuniceras i form av sekvenstyper (ST). Den är, liksom för andra bakteriearter, mindre diskriminerande än PFGE och lämpar sig därför bättre för globala fylogenetiska studier. MLVA är också en sekvensbaserad metod och snabbare än MLST. Den har ännu inte prövats i tillräcklig omfattning för att man ska kunna rekommendera den som alternativ till PFGE.
Det finns i nuläget (år 2012) ingen konsensus internationellt om vilken metod som är mest lämpad för epidemiologisk typning av VRE, varken för E. faecalis eller för E. faecium. Det vore önskvärt med en sekvensbaserad metod (liknande spa-typning för MRSA) som är överlägsen i snabbhet och som ger resultat som enkelt kan kommuniceras till omvärlden.
Fram till 2006 anmäldes till SMI i genomsnitt 26 nya fall per år från hela landet. Under andra halvåret 2007 ökade antalet anmälda VRE-fall signifikant. Även 2008 ökade antalet anmälda fall kraftigt, men följdes av färre antal anmälningar under 2009 och 2010. Smittskyddsinstitutet anser att det är viktigt att Sverige kan behålla sin i internationell jämförelse relativt gynnsamma situation avseende VRE. Därför bör en samlad nationell kunskap om genetisk likhet mellan identifierade isolat som påvisar aktuell smittspridning prioriteras. Detta för att skapa underlag till såväl riktade preventiva insatser som till utvärdering av insatserna.
Sammanfattning av avsnitt 5
Epidemiologisk typning har i huvudsak tre syften;
- vid utredning och kontroll av VRE-utbrott på lokal nivå
- för nationell samording av kunskapen om utbrotten för att få en överblick över epidemiologin
- vid utvärdering av preventiva insatser
- typning av VRE utförs i dagsläget huvudsakligen med PFGE.
- Misstänkta klonala utbrott bör verifieras med PFGE.
- för nationell överblick av pågående VRE-spridning inom landet bör isolat från samtliga nyupptäckta fall skickas till Smittskyddsinstitutet för epidemiologisk typning. Återkoppling till insändande laboratorium och till ansvarig smittskyddsenhet bör ske inom 14 dagar.
Bilagor till VRE-diagnostiken
REFERENSER
- 1. Föreningen för Medicinsk Mikrobiologi vid Svenska Läkaresällskapet och Smittskyddsinstitutet. Referensmetodik för laboratoriediagnostik vid kliniskt mikrobiologiska laboratorier utgiven. Artikel Enterokocker och streptokocker-minimikriterier vid speciesbestämning
- 2. Pendle S, Jelfs T, Olma T, Su Y, Gilroy N, Gilbert GL. Difficulties in detection and identification of Enterococcus faecium with low-level inducible resistance to vancomycin, during a hospital outbreak. Clinical Microbiology and Infection. 2008 Sep; 14(9):853-7.
- 3. Snyder JW, Munier G, Johnson C. Direct Comparison of the BD PhoenixTM Automated Microbiology System with the Microscan Walkaway for Identification and Antimicrobial Susceptibility Testing of Staphylococci, Enterococci and Antimicrobial Susceptibility of Streptococci. Presented at the 106th General Meeting of the American Society for Microbiology (ASM), Orlando, FL, 2006.
- 4. Kobayashi I, Muraoka H, Iyoda T, Nishida M, Hasegawa M. Yamaguchi K. Antimicrobial susceptibility testing of vancomycin-resistant Enterococcus by the VITEK 2 system, and comparison with two NCCLS reference methods. J Med Microbiol. 2004 Dec; 53 (Pt 12):1229-32.
- 5. Ieven M, Vercauteren E, Descheemaeker P, van Laer F, Goossens H. Comparison of direct plating and broth enrichment culture for the detection of intestinal colonization by glycopeptide- resistant enterococci among hospitalized patients. J Clin Microbiol. 1999 May; 37(5): 1436-40.
- 6. Isenberg HD, Goldberg D, Sampson J. Laboratory studies with a selective Enterococcus medium. Appl Microbiol. 1970 Sep; 20(3): 433-6.
- 7. Palladino S, Kay ID, Costa AM, Lambert E J, Flexman JP. Real-time PCR for the rapid detection of vanA and vanB genes. Diagn Microbiol Infect Dis. 2003 Jan; 45(1):81-4.
- 8. Dahl KH, Simonsen GS, OlsvikO, Sundsfjord, A. Heterogeneity in the vanB gene cluster of genomically diverse clinical strains of vancomycin-resistant enterococci. Antimicrob Agents Chemother. 1999 May; 43(5):1105-10.
- 9. Depardieu F, Perichon B, Courvalin P. (2004). Detection of the van alphabet and identification of enterococci and staphylococci at the species level by multiplex PCR. J Clin Microbiol. 2004 Dec; 42(12): 5857-5860.